研究の目的
新規遺伝子発現解析法SuperSAGE法を活用して、イネ-いもち病菌相互作用を解析し、いもち病菌の感染機構、イネの抵抗性機構を明らかにする。
研究項目及び実施体制(◎は研究代表者)
- SuperSAGE法によるイネ-いもち病菌相互作用の遺伝子発現解析
(◎寺内良平、松村英生/ (財)岩手生物工学研究センター) - SuperSAGE-RNAi法の確立
(松村英生/(財)岩手生物工学研究センター) - イネおよびいもち病菌におけるTILLING法の確立
(神崎洋之、藤澤志津子/(財)岩手生物工学研究センター) - イネの形質転換による遺伝子機能解明
(神崎洋之、藤澤志津子/(財)岩手生物工学研究センター) - イネで同定された遺伝子の双子葉植物における機能解明
(伊東明子/(財)岩手生物工学研究センター) - いもち病菌の遺伝子破壊
(齋藤宏昌、藤澤志津子、伊東明子/(財)岩手生物工学研究センター) - タンパク質相互作用の解析
(神崎洋之/(財)岩手生物工学研究センター)
研究の内容及び主要成果
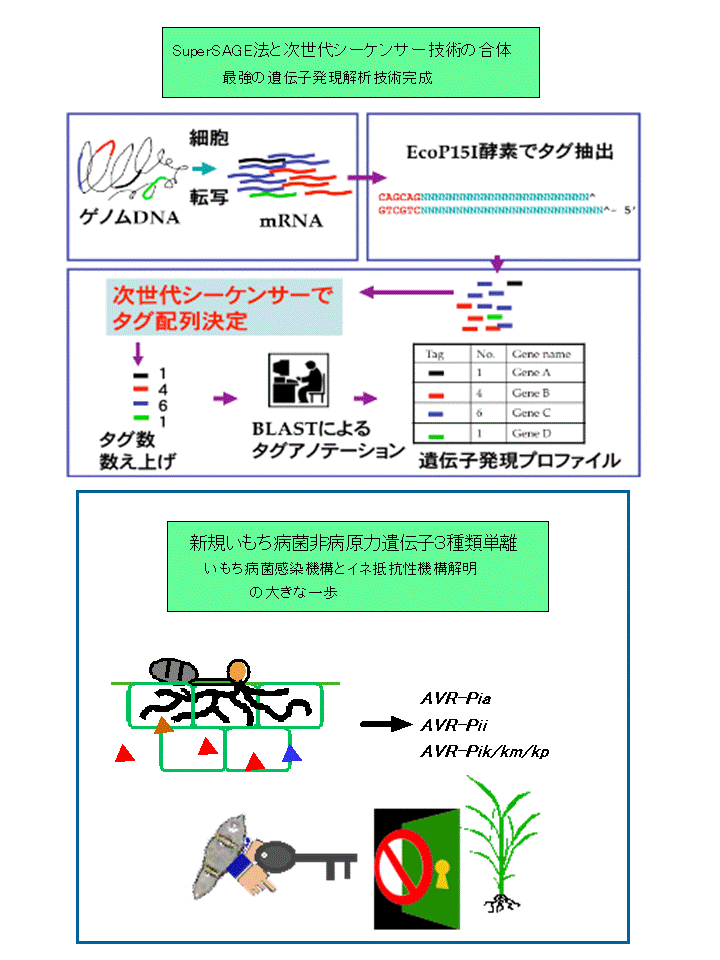
- SuperSAGE法と次世代シーケンサー技術を合体して、現時点で最強の遺伝子発現解析技術を作り上げた。
- 日本産いもち病菌ゲノム配列を解読し、新規非病原力遺伝子AVR-Pia,AVR-Pii,AVR-Pik/Pikm/Pikpを単離
- いもち病菌から分泌されるエフェクタータンパク質を多数同定、イネとの相互作用解明に着手
- 北日本で重要な水稲品種「ひとめぼれ」突然変異系統、約8500系統を作出。
見込まれる波及効果
次世代シーケンサーを活用したSuperSAGE解析により、農林水産物の遺伝子発現解析が飛躍的に簡便迅速化された。いもち病菌新規非病原力遺伝子の単離により、迅速なレース判定が可能になり、マルチラインに使用するイネ抵抗性遺伝子選択が容易になった。いもち病菌エフェクターの機能解明により、植物耐病性機構の新パラダイムが呈示できる。
主な発表論文
- Matsumura, H., et al.: SuperSAGE-array: The direct use of 26bp transcript tags in ologonucleotide arrays for deep and genome-wide gene expression analysis. Nature Methods 3 : 469-474 (2006)
- Takahashi, Y., et al. : A high-throughput screen of cell-death-inducing factors in Nicotiana benthamiana identifies a novel MAPKK that mediates INF1-induced cell death signaling and non-host resistance to Pseudomonas cichorii. Plant J. 49: 1030-40 (2006)
- Terauchi, R., et al.: A multi-faceted genomics approach toward understanding Magnaporthe-rice interactions. In: Biology of Molecular Plant-Microbe Interactions. (Lorito, M. ed.) Vol. 6 (2008)
- Takahashi K., et al.: Serine palmitoyltransferase, the first step enzyme in sphingolipid biosynthesis, is involved in non-host resistance. Molecular Plant-Microbe Interact. 22:31-38 (2009).
- Yoshida K., et al.: Association genetics reveals three novel avirulence genes from the rice blast fungal pathogenMagnaporthe oryzae. The Plant Cell. 21: in press (2009)
研究のイメージ