開発の社会的背景
自然界において、紫外線などでDNAが切断されることにより遺伝子のDNA配列が変化することがあり、こうした突然変異が作物の生育特性や品質などの性質の変化を導きます。人類は、このようにして得られた作物の中から有用な性質を品種改良に利用してきました。
放射線の照射や薬品処理等により突然変異を人為的に起こす方法も用いられていますが、目的の性質を持つ変異個体を取得するには偶然に頼らなければならず、多数の個体を長い年月をかけて栽培し、選抜する必要があります。
近年、この問題を克服するためにゲノム編集技術が活用されています。ゲノム編集技術の中でも「欠失型ゲノム編集」と呼ばれる技術は、特定のDNA配列を狙って切断することで、目的の遺伝子に変異を起こさせることができます。そのため、新たな変異を効率的に導入し、目的の性質を持つ個体を取得することができます。現在では多くの遺伝子の機能が明らかになってきていることから、この技術を用いて、トマトに含まれるGABA(血圧上昇抑制効果があるアミノ酸)の生成に関わる遺伝子に変異を起こすことでGABA含有量を高めたトマトなどが開発されています。
研究の経緯
ゲノム編集作物を作出する際には、多くの植物では目的のDNAを切断するための酵素を作る遺伝子(ゲノム編集酵素遺伝子)を一度細胞内に導入する方法がよく用いられます。ゲノム編集酵素遺伝子は外来の遺伝子(外来DNA)であるため、この遺伝子を有すると遺伝子組換え体となります。そこで、目的の作物が作出された後は、交配や選抜を経てゲノム編集酵素遺伝子を有しない作物を取得します(図1)。このようにして作出されたゲノム編集作物を利用するためには、ゲノム編集酵素遺伝子などの外来DNAがきちんと取り除かれていることを確認する必要があります。
その検出法として、農研機構では世界で初めて、次世代シーケンサーを用いて解読された膨大なDNA配列データの解析と統計検定を組み合わせて、ゲノム編集酵素遺伝子のDNA配列を効率的かつ高精度に検出できるk-mer法(図1)を開発し、2020年から解析ツールを公開してきました。
しかし、k-mer法解析ツールを使用するためには、データ解析と統計検定を4つのステップに分けて実行していく必要があり(図2)、手順が煩雑でゲノム解析を専門とした研究者が不可欠でした。また、統計検定を実施すると偽陽性が検出されることがありますが、k-mer法解析ツールには偽陽性を抑える機能がありませんでした。そこで今回、これまでの解析ツールを改良して操作を簡便にし、より正確に判別できるようにした解析ツール "GenEditScan"を開発しました。
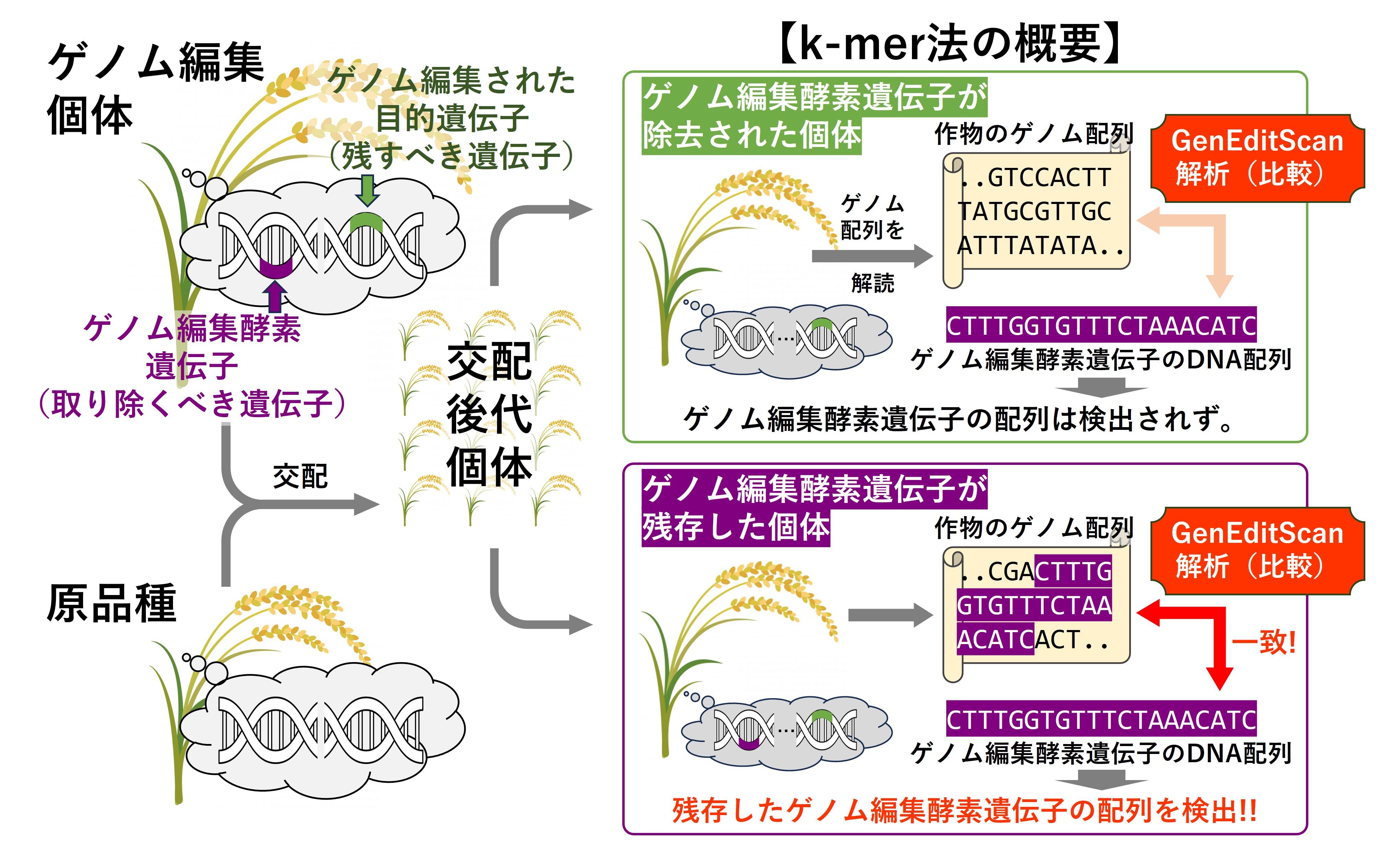 図1:ゲノム編集個体に残存しているゲノム編集酵素遺伝子を取り除く過程の例とk-mer法の概要
図1:ゲノム編集個体に残存しているゲノム編集酵素遺伝子を取り除く過程の例とk-mer法の概要
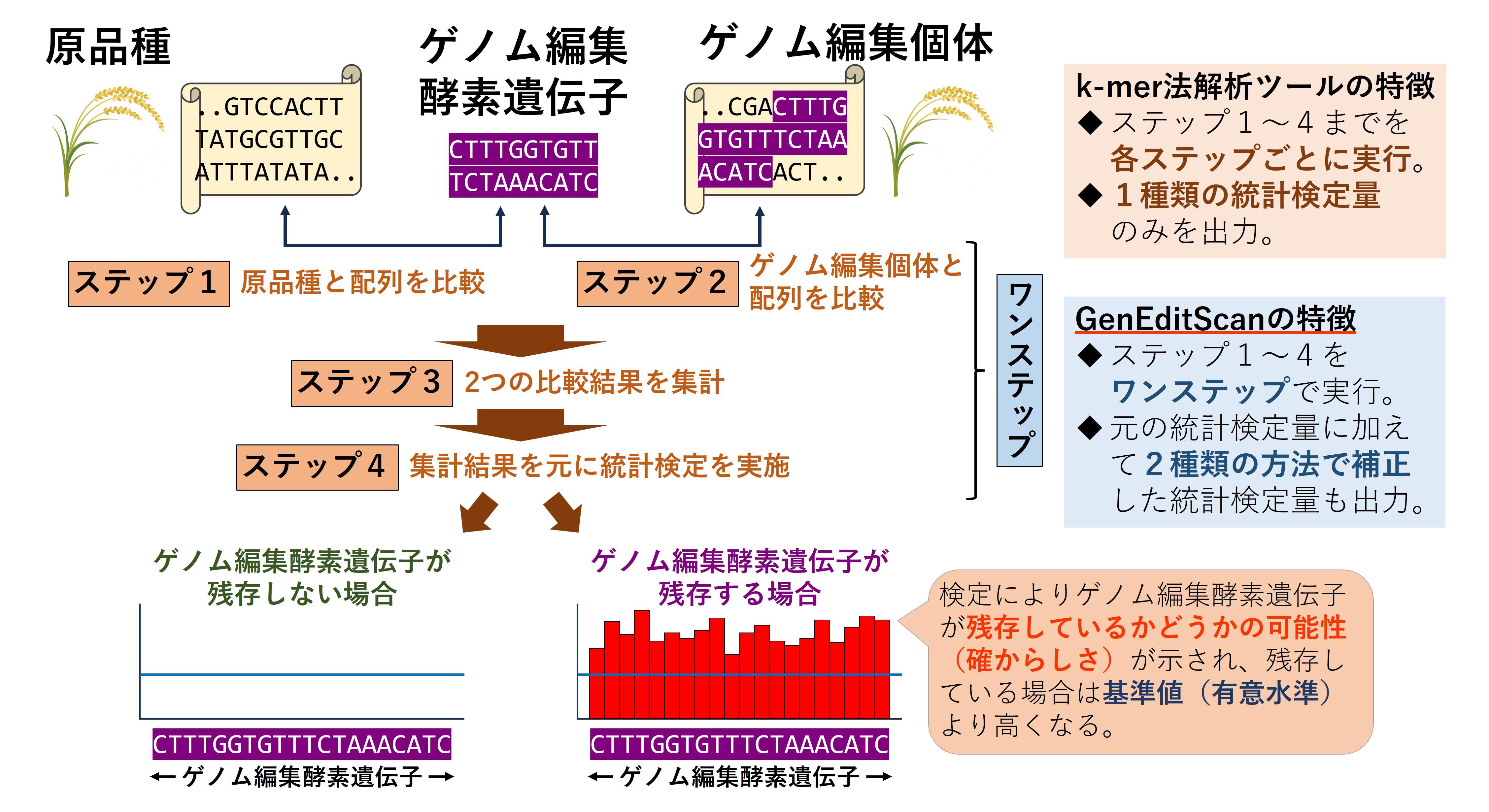 図2:k-mer法解析ツールとGenEditScanの実行手順と特徴
図2:k-mer法解析ツールとGenEditScanの実行手順と特徴
研究の内容・意義
GenEditScanは、次世代シーケンサーで解読したゲノム編集個体および原品種のゲノム情報と、ゲノム編集酵素遺伝子の塩基配列情報を入力するだけで、ワンステップでゲノム編集酵素遺伝子の有無を解析できるようプログラムに工夫を施しました(図2)。また、解析の過程で生じる統計検定結果を補正5)することにより、特定の条件でシミュレーションを実施した際の偽陽性を3分の1未満に抑えることができ、より正確に判別できるようになりました。これらは、利用者の知識や経験によらず、どなたが使用しても簡便に同じ結果が得られます。
一例として、実際にゲノム編集技術によって作成されたバレイショを対象にGenEditScanによる外来DNAの検出を行った結果を示します(図3)。外来DNAが残存していることが別の実験によってあらかじめ確認されているゲノム編集個体では、GenEditScanでも外来DNAを検出できる(図3上)一方、残存していないことが実験的に確認されている個体では、GenEditScanでも検出されませんでした(図3下)。これらの結果から、GenEditScanにより外来DNAの有無を正確に判別できることが示されました。
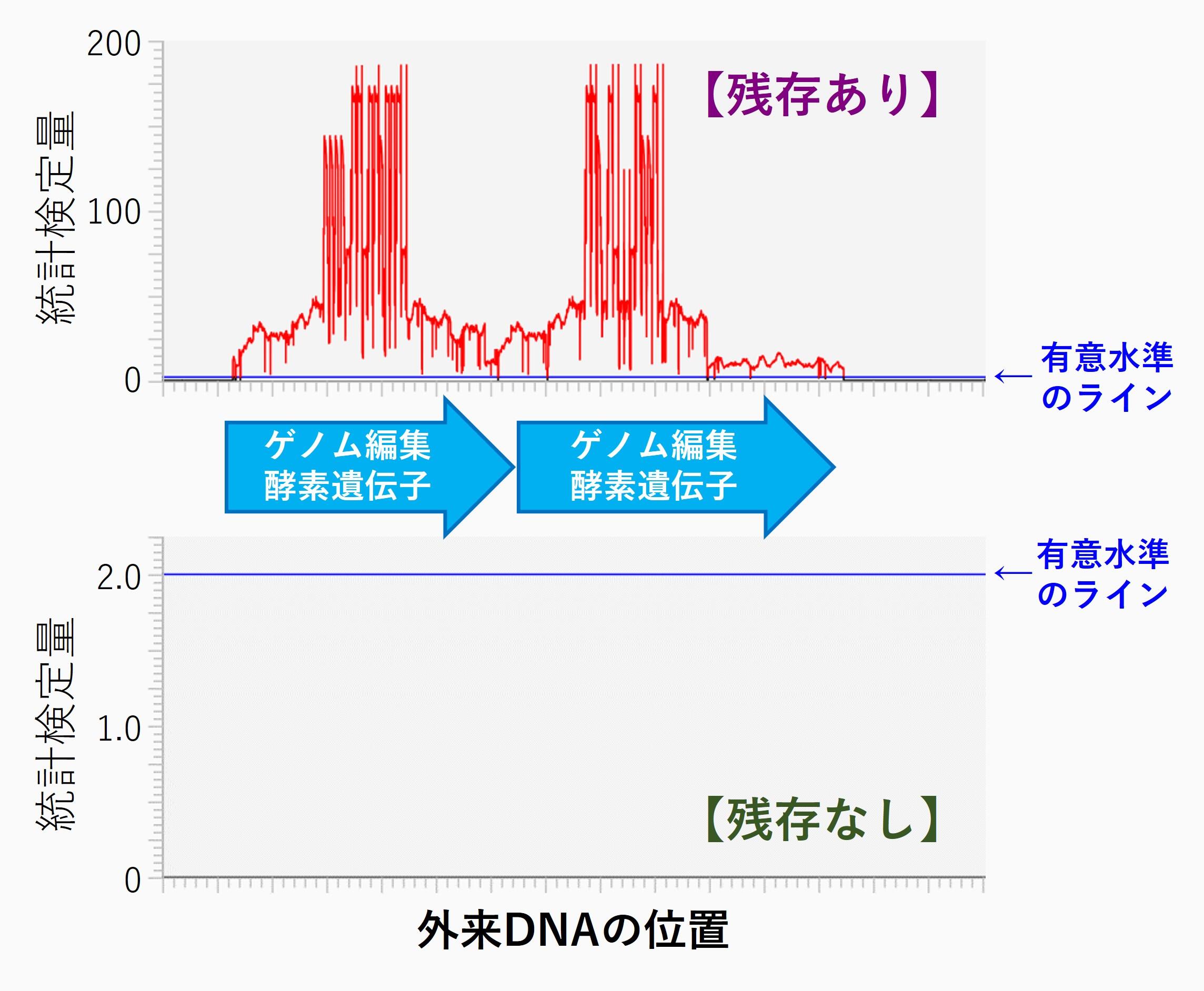 図3:GenEditScanの利用例
図3:GenEditScanの利用例
Yasumoto and Muranaka (2023) (https://doi.org/10.1038/s41598-023-38897-x)で公開されたゲノム配列(DDBJ:DRA015756)をGenEditScanで解析して作成したグラフ。グラフの横軸は外来DNAの位置、縦軸は統計検定量(FDR制御)を示しています(青色の横線は有意水準1%のライン)。外来DNAが残存している場合、上図のように、外来DNAが残存している領域に通常ピークが検出されます(赤線)。一方、ゲノム編集酵素遺伝子が残存していない場合は、下図のように有意水準(青色の横線)を超えるピークは基本的に検出されません。
今後の予定・期待
GenEditScanの前身である解析ツールは、ゲノム編集作物を研究段階において屋外で栽培するための官庁への情報提供の中で、ゲノム編集作物に外来DNAが残存していないことを確認する手法として、すでに3件の利用実績(イネ、ジャガイモ、コムギ)を有しています。GenEditScanの利用には専門的な知識と経験を要する実験的な作業は必要なく、ゲノム配列情報を取得できればどのような作物でも利用可能です。今後はゲノム編集作物に外来DNAが残存していないことを確認する新たな手法として、GenEditScanが活用され、ゲノム編集技術を用いた効率的な有用作物開発に貢献できるものと期待されます。
農研機構高度分析研究センターでは、上記のようなプログラムの開発のみならず、プログラムの構築や研究・解析の支援、機器・施設利用の提供、またそれらを糸口とした共同研究開発を積極的に推し進めています。
用語の解説
- 次世代シーケンサー
- 生物のゲノム情報を一度に大量に解読できる専用機器のこと。イルミナ社の次世代シーケンサーでは最大で一度に16兆塩基のゲノム配列を解読できます。例えば、ヒトの塩基対数は約32億ですので、これは5,000人分の塩基対数に相当します(16兆÷32億=5,000)。[ポイントへ戻る]
- ゲノム
- 生物の遺伝情報の全体のこと。「アデニン(A)」「チミン(T)」「グアニン(G)」「シトシン(C)」の4種類の塩基から構成されるデオキシリボ核酸(DNA)でコードされており、すべての生物に存在します。遺伝子はDNAのうちその生物の性質や特徴などに大きく関与する部分をいいます。[ポイントへ戻る]
- k-mer法
- 次世代シーケンサーで解読されたゲノム編集個体及び原品種のゲノム配列データにおいて、長さkの外来DNAの配列(k-mer)と一致する配列の検出数をカウントし、両者のカウント数を統計学的に比較することで、ゲノム編集個体中に外来DNAが含まれているか判断する方法のこと。外来DNAの長さが20塩基以上の場合、ほぼ100%検出できます。[ポイントへ戻る]
- 偽陽性
- 統計検定において、実際には有意な効果や差が存在しないにもかかわらず、誤って有意と判断してしまうことを指します。統計検定を繰り返し行う多重比較では、検定回数が増えるほど、少なくとも1つの検定で誤って有意と判断する確率(誤検定率)が上昇し、誤った結論を導くリスクが高まります。そのため、FDR(False Discovery Rate、偽発見率)制御やBonferroni法などの補正法が用いられます。[概要へ戻る]
- 偽陽性を抑える手法
- GenEditScanでは2つの方法で偽陽性の検出を減らしています。1つ目はFDR制御で、統計検定を繰り返し行う際に、有意とされた結果の中で偽陽性の割合を一定の値以下に制御しつつ、実際に存在する効果や差を見落とすリスクを最小限に抑えるようにバランスを取るための方法です。GenEditScanでは、FDR制御の代表的な方法であるBenjamini-Hochberg法を使って統計検定結果の補正を実施しています。2つ目はBonferroni法です。統計検定を繰り返し行う多重比較における補正法の1つで、全体の有意水準を維持するために各検定の有意水準を全体の検定数で割る方法です。これにより偽陽性を抑えることができますが、実際に存在する効果や差を見落とすリスクが高まるため、GenEditScanでは2種類の補正方法を取り入れています。[研究の内容・意義へ戻る]



