開発の社会的背景
大豆は世界的には油脂生産や家畜飼料に利用される重要な作物ですが、日本では昔から豆腐、味噌、納豆、醤油などに使われ、食文化と深く関わる大切な食材です。国産大豆は高タンパク質や子実外観の美しさなど、食品向けの品質の良さが知られていますが、収量が低く不安定なことが課題です。そこで農研機構は、収量の高い米国品種と日本品種を交配し、多収で豆腐向けの新品種群「そらシリーズ」(「そらみのり」、「そらたかく」、「そらみずき」、「そらひびき」の4品種)を育成し、普及を開始しました。「そらシリーズ」は従来品種の1.2倍以上の収量を実現していますが、米国品種と日本品種の双方の特長を完全には併せ持っていません。そのため、双方の特長をゲノムレベルで理解し、科学的な知見に基づいた育種がより重要になっています。
研究の経緯
これまでの研究から、日本の大豆品種は世界の主要品種と比べて、ゲノム塩基配列に多くの違いがあることが示唆されていました。しかし、日本品種のゲノム全体の詳細な塩基配列情報や、複数の代表的な品種の詳しいゲノム情報が不足していたため、日本品種に特有なゲノム構造を十分に理解できていませんでした。そこで今回、日本品種の代表として「エンレイ」や「フクユタカ」などの全ゲノムを最新のロングリードDNAシークエンサーを使って詳しく調べました。さらに、ゲノム構造の特徴を客観的に評価するための新しい情報解析(インフォマティクス5) )手法を開発し、米国品種とのゲノム配列や構造の比較解析により日本品種の特性をより深く理解することを目指しました。
研究の内容・意義
日本品種を含む11品種の全ゲノム情報の取得 ゲノムアセンブリ6) という)を新たに取得しました。なかでも「エンレイ」については国産大豆の基準品種として、約6万の遺伝子の位置を明らかにした参照ゲノム情報(リファレンスゲノム7) ともいう)を整備しました。例えばエンレイゲノムでは、どれだけの遺伝子を網羅しているかを表すBUSCO5指数は99.8%を記録し、世界最高レベルの精度であることが確認されました。さらに、エンレイ以外の10品種についても、それぞれ6万程度の遺伝子の位置を明らかにし、リファレンスゲノムとして整備しました。日本品種と海外品種のゲノム構造の違いをパンゲノム解析により解明 パンゲノムグラフ8) 」に統合し、安価に取得可能なショートリードデータ9) をゲノム構造変異解析に用いることに成功しました。これにより、数百系統規模の大豆品種のゲノム構造変異解析にかかる費用を、ロングリードシークエンスを利用する場合に比べて、1/10程度に減らすことができます。明らかになった日米品種の大きな違い 図2 )。また、ゲノム構造変異だけでなく、一塩基多型などの微細なゲノム配列変化も明らかになりました。特に、日米品種間ではそれらの違いが顕著でした(図3 )。例えば、莢のはじけにくさや病害抵抗性、子実の大きさや形状を決める遺伝子について、ゲノム構造や配列の違いが見つかりました。日本独自の高温多湿な気候風土や独特な大豆食文化が、長い年月を経て日本品種のゲノムの違いに影響した可能性があると考えられます。
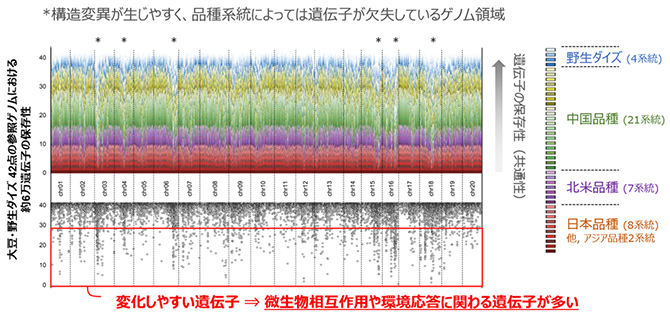
図2 日本・海外大豆のゲノムにおける遺伝子構造変異の解析 由来の異なる大豆・野生大豆系統のゲノムを、独自に開発したゲノム情報解析プログラム「Asm2sv」で調べ、全ての遺伝子についてどのくらい共通しているかを計算しました。その結果を、縦積棒グラフ(上)と点グラフ(下)として示しています。双方とも、縦軸の数値が低い場合は、異なる品種系統ゲノム間で遺伝子が保存されていない傾向を示します。解析の結果、日本と海外品種の間で構造変異が生じている染色体の領域がいくつも見つかりました。これらの領域には、微生物との相互作用や、異なる栽培環境への適応に関わると考えられる遺伝子が多く含まれていました。(図は論文から改編・転載)
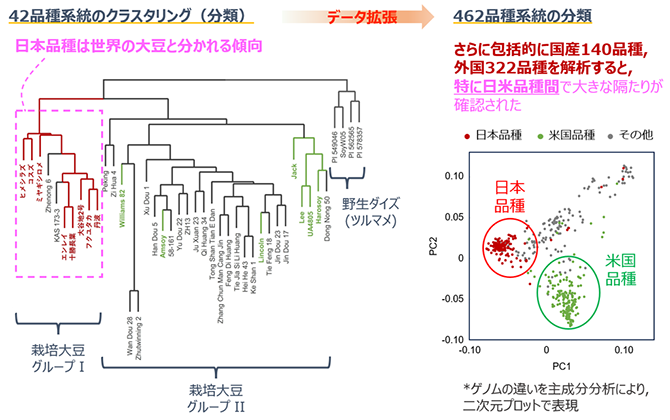
図3 ゲノムデータによる日本と海外大豆の分類 図2 で得られたゲノム構造変異のデータをもとに、大豆42品種のグループ分け(クラスタリング)した結果を左図に示しています。また、日本140品種、海外322品種のゲノム配列の違いを主成分分析で可視化した結果を右図に示しています。右図は、DNA配列のうち、一塩基ごとの違いに基づきます。これらの解析から、日本品種(赤色)は海外品種と明確に区別されることがわかり、特に米国品種(緑色)とゲノム構造に大きな違いがあることが明らかになりました。(図は論文から改編・転載)
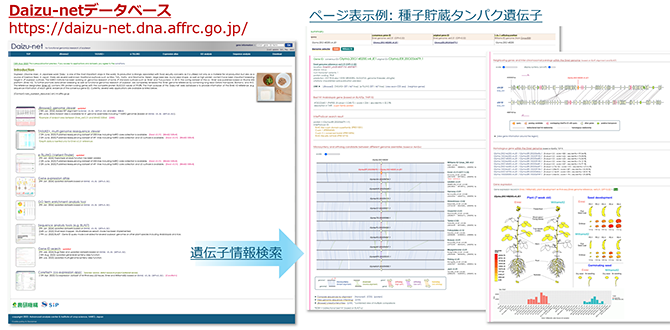
Daizu-net (ゲノム・遺伝子情報検索データベース)の開発と公開(図4 ) 古倍数性10) 」も考慮した遺伝子解析機能を備えており、主に国内の研究者の育種や研究活動を強力にサポートすることが期待されています。
図4 Daizu-netデータベースの遺伝子情報ページの例 このデータベースでは、本研究にて詳細に解析した日本大豆の基準品種「エンレイ」の遺伝子情報やその他のゲノムデータをウェブブラウザ上で簡単に確認できるようになっています。さらに、異なる品種ゲノムで同じ働きをもつ遺伝子(オルソログ)の対応関係や、同祖遺伝子(異なる染色体にあっても、もともと同じ遺伝子から分かれたと考えられる遺伝子)の情報も見ることができます。加えて、遺伝子がどの部分でどれくらい働いているかを示す「遺伝子発現アトラス」も閲覧可能です。
今後の予定・期待
本研究によって、日本の主要な大豆品種の全ゲノム情報が明らかになったことで、日本や米国など各国の品種の特徴を決める遺伝子やゲノム構造の違いの理解が進み、日本品種の収量性を改善しつつ、子実品質を損なわない新品種の育成が進むと期待されます。また、今回確立されたゲノム情報処理やデータベース構築の技術(インフォマティクス解析技術)を使えば、日本品種だけでなく、世界中の大豆をより広く、そして詳しく調べることが可能となります。こうした解析によって、各国の栽培環境や用途に応じて、どのような遺伝子の変化が育種の過程で起こったのかを客観的に分析できるようになります。そして、これらの情報を蓄積することで、温暖化や省資源(肥料や農薬の削減)など農業のおかれる環境の変化にも迅速に対応して品種を開発できると考えています。
用語の解説
ロングリードDNAシークエンサー
生物の体は、設計図のような情報をもとに作られています。この設計図は「DNA」と呼ばれ、細胞の中の「核」にある「染色体」に含まれています。各染色体に1本ずつDNA分子があり、各DNA分子は4種類の塩基という小さな単位がつながったとても長い鎖のようになっています。大豆の染色体数は全部で20本あり、合計約10億の塩基から成り立っています。このような塩基の連なりを「塩基配列」もしくは「DNA配列」と呼び、それらの総体を「ゲノム」と呼びます。
ロングリードDNAシークエンサーは、そのようなゲノムDNA配列を読むための最新の機械の一つです。従来の方法(ショートリードシークエンサー、下記参照)ではDNAを細かく切ってから読んでいましたが、ロングリードDNAシークエンサーでは長いままのDNAを一気に読むことができます。
そのため、より正確で詳しいゲノム配列情報を手に入れることができます。ただし、ロングリードDNAシークエンサーはショートリードシークエンサーに比べて、研究費用が高額になる弱点があります。
[ポイントへ戻る]
全ゲノム情報
1)に述べたように、大豆ゲノムは約10億の塩基で構成される20本の染色体で成り立っています。大豆ゲノム配列には約6万個の遺伝子が含まれていて、それぞれの遺伝子の塩基の並び方にしたがって、タンパク質が作られます。そして、それらのタンパク質が様々に働くことで、種子が発芽し、生長し、そして種を生み出します。品種ごとの特徴(例えば、子実の品質や収穫量、異なる栽培環境への適応など)も、ゲノムDNA配列の違いによって生じます。
ゲノム研究においては、全DNA配列とそこに含まれる全遺伝子セットの双方を指して、全ゲノム情報とよびます。
[ポイントへ戻る]
ゲノム構造変異
大豆ゲノムには約6万個の遺伝子がありますが、その内容は品種ごとに少しずつ異なります。違いには、塩基(DNAの文字)のわずかな違いだけでなく、大きな単位でDNA配列が入れ替わったり、無くなったりするような変化があります。
こうした変化により、品種によっては遺伝子がまるごと欠失したりしますが、このような構造的な変化を総称して、ゲノム構造変異(英語: genomic structural variation)とよびます。
これまでは一塩基多型など小さな違いしか詳しく調べられませんでしたが、今回の研究では最新のロングリードDNAシークエンサーに独自のゲノム情報解析技術を組み合わせることにより、大きな違いも高い精度で多くの品種間で比べられるようになりました。
[概要へ戻る]
機械化適性
大豆を大規模に栽培するには、種播き、収穫、脱穀等を機械で行う必要があります。担い手不足により1つの農家の栽培面積が大きくなりつつあることから、機械化の重要性は年々高まっています。
機械収穫を行うためには、大豆の植物体が容易には倒れないこと、機械が触れても莢が弾けにくいこと、などの特長が必要で、これらのような特長をまとめて機械化適性といいます。米国の大豆品種はこうした機械化に向いた特性が改良されています。
[図1へ戻る]
インフォマティクス
ゲノムや遺伝子の情報など、生体情報を収集してデータ化し、スーパーコンピュータ等の計算機を用いて分析する情報処理技術のことです。塩基配列を調べたり、ゲノムの構造を比較したり、遺伝子の機能を予測したり、といったことが、正確かつ効率よく行えるようになります。育種分野では品種の遺伝情報を理解し、迅速に改良を加えるうえで、とても重要な技術です。
[研究の経緯へ戻る]
ゲノムアセンブリ
ロングリードDNAシークエンサーを使っても、染色体のDNAを最初から最後まで一気に読むことはまだできません。そこで、断片的に得られたDNAの配列データをパズルのように組み合わせて、本来の染色体に近い形に並べ直す作業が必要です。このようにして作られた染色体DNA配列データをゲノムアセンブリといいます。
[研究の内容・意義へ戻る]
リファレンスゲノム
ゲノムアセンブリと、ゲノム上に存在する遺伝子の位置情報の双方を合わせたデータを、リファレンスゲノムといいます。全ゲノム情報は、リファレンスゲノムと同義で用いられることがあります。
[研究の内容・意義へ戻る]
パンゲノムグラフ
7)で述べたリファレンスゲノムは、それぞれの品種系統の全ゲノム情報を1つずつ解読してデータ化したものです。しかしながら、それぞれの品種のゲノムの間では、遺伝子が丸ごと欠失したり、重複したりと、多様なゲノム構造変異が起こり得ます。そのような多様なゲノム構造変異を統合して、単一のデータとして表現したものが、パンゲノムグラフ(英語: pangenome graph)です。パンゲノムグラフでは、ゲノム構造変異は異なるパス(通り道)として表現されます。すなわち、あるゲノム領域において複数の構造変異が存在すれば、その数だけパスが増えます。一度、パンゲノムグラフが完成すると、次の9)で述べる安価なショートリードデータを利用して、数百系統規模のゲノム構造変異解析を実施できるようになります。
[研究の内容・意義へ戻る]
ショートリードデータ
ロングリードシークエンサーが出現する前から存在する、従来からのゲノム配列解析手法がショートリードシークエンスです。ショートリードシークエンサーでは、150塩基程度の非常に短い塩基配列データを取得して解析しますが、ロングリードシークエンスに比べて1/10程度の研究費用で安価にデータを得ることが可能です。
一塩基多型などの小さなゲノムの違いは、それぞれの品種で取得されたショートリードデータを、7)で述べたリファレンスゲノムに照らし合わせて解析することで調べることができます。
他方で、8)で述べたパンゲノムグラフを参照ゲノムとして用いることで、ロングリードシークエンスを実施せずにショートリードデータのみで、大規模かつ安価にゲノム構造変異を調べることができます。
[研究の内容・意義へ戻る]
古倍数性
大豆では、約1,300万年前にゲノム全体が二倍に重複したと考えられています。その後、重複した遺伝子の一部は使われなくなったり消えたりしましたが、その痕跡がいまもゲノムの中に残っています。このような現象を「古倍数性」といいます。大豆の中にはとても似たDNA配列の遺伝子がたくさんあり、それぞれの遺伝子を区別したり、どれが実際に働いているかを判断するのが難しくなっています。
[研究の内容・意義へ戻る]
発表論文
Yano R., Li F., Hiraga S., Takeshima R., Kobayashi M., Toda K., Umehara Y., Kajiya-Kanegae H., Iwata H., Kaga A., Ishimoto, M. (2025) The genomic landscape of gene-level structural variations in Japanese and global soybean Glycine max cultivars. Nature Genetics 57: 973-985. https://doi.org/10.1038/s41588-025-02113-5
研究担当者の声
高度分析研究センター矢野 亮一
本研究では、約10億個のDNA塩基から構成される大豆ゲノムを全ての染色体で解読し、リファレンスゲノムを複数品種で構築しました。特に、それら品種ゲノムの間で生じているゲノム構造変異に注目しましたが、大豆ゲノムはイネやシロイヌナズナなどのモデル植物とは異なって、過去に一度、全染色体が倍増してランダムに組み合わさった複雑な構造を持ちますので(古倍数性)、従来の解析メソッドをそのまま当てはめることが困難でした。本研究では、Asm2svと名付けた新規のゲノム構造変異解析プログラムを開発し、この問題に挑戦しましたが、プログラミング作業が最も大変だった時は気付いたら夜が明けていたこともしばしばあり、今回、そのような研究の取り組みが論文としてNature Geneticsという世界最高峰の科学誌に掲載されたことを非常に嬉しく思います。