開発の社会的背景
キク(栽培ギク)は我が国の切り花生産本数の約3分の1を占め、花き生産の基幹となる品目です。我が国では年間約19億本が消費されますが、お盆やお彼岸の需要期の供給不足を背景に、近年は赤道直下の高地に位置する新興花き産地から我が国への輸出が急増し、国内市場流通に占める輸入品のシェア増大が続いています。需要期の供給不足を補い、国産シェアを奪還するため国内の増産が求められており、生産性が高く、病害虫に強い品種開発が急務となっています。現在のキクの品種開発は、育種者の経験に基づいて行われていますが、DNAマーカーなどのゲノム情報を活用することで効率的な品種開発が可能となります。しかしキクは同質六倍体で遺伝様式が複雑なためDNAマーカーの開発がきわめて困難でした。
研究の経緯
花の形態や生育開花習性がキクと同様である二倍体野生種のキクタニギクをモデル植物として用い、キクの同質六倍体の問題を解決することを目指してきました。これまで、キクタニギクの全ゲノムの解読や遺伝子地図(連鎖地図)を作成し、キクの育種基盤として整備してきました(2019年2月4日、かずさDNA研究所、農研機構ほかプレスリリース)。
研究の内容
キクのDNAマーカーを効率的に開発する手法を確立しました。本手法では、以下の方法でDNAマーカーを開発します。
- ddRAD-seq解析4)によって、キクのゲノム配列の断片情報を取得します。
- キクタニギクのゲノムを参照配列として、キクのゲノム配列断片の対応する部分を並べ、DNA多型5)を見つけるとともに、その座乗位置を明らかにします。
- 座乗位置の情報から6ゲノムのうち1ゲノムだけに座乗するDNA多型だけを選抜します。
- 関連解析により有用な性質と相関のあるDNA多型を同定し、DNAマーカーを開発します。複数ゲノムに座乗するDNA多型は遺伝が複雑で解析が困難なため、1ゲノムだけに座乗するDNA多型をマーカーとして用いることによって、同質六倍体においても関連解析を利用したDNAマーカー開発が可能になります。
この手法を用いて花弁色に関するDNAマーカー開発を試みました。その結果、既知の花弁色決定遺伝子(CmCCD4a遺伝子)の近傍にDNAマーカーを同定できたことから、手法の有効性が確認されました。
今後の予定・期待
本手法はキクの品種開発に関連する機関でのマーカー開発の際に活用が見込まれます。今後は、DNAマーカーを使うことによって、形質による選抜に要する時間と手間の軽減、複数の有用形質を導入する時間と手間の軽減など、ゲノム情報を活用したキクの効率的な品種開発が可能となります。
用語の解説
- 同質六倍体
- 生物が生存に必要な最小限の染色体のセット(ゲノム)を何セット持っているか示す概念を倍数性といいます。この倍数性に基づいた生物の分け方が倍数体です。多くの生物は二倍体で2セットのゲノムを持っていますが、六倍体では6セットのゲノムを持っています。 六倍体には、コムギのように3種の異なる植物種由来の6セットのゲノムを持つ異質六倍体と、キクのように、ほぼ同じ6セットのゲノムをもつ同質六倍体があります。同質倍数体は、二倍体や異質倍数体に比べて複雑な遺伝様式をとります。
- DNAマーカー
- 遺伝子の目印となるDNA配列。導入したい形質に関わる遺伝子をDNAマーカーの有無で確認して個体を選抜することが可能となります。さらに、従前の形質(例えば、「背が高い」、「色が濃い」といった見た目の性質)を対象とする選抜とは異なり、環境による影響を受けないので安定して利用できます。
- 関連解析
- 形質の違いと相関のあるDNAマーカーを統計的に検出する手法。ゲノムワイド関連解析ともいわれます。これまで、ヒトの疾患にかかわる遺伝子の同定や疾患の機構の解明に貢献してきましたが、近年は作物においても応用されています。
- ddRAD-seq解析
- 2種類の制限酵素でゲノムを切断し、両端が別々の制限酵素で切断された断片のみを次世代シーケンス技術によって解析するもので、ゲノムを再現性良く読むことができ、塩基配列の違いを容易に比較することができます。
- DNA多型
- ゲノムDNA中の塩基配列にみられる変異。
発表論文
Katsuhiko Sumitomo, Kenta Shirasawa, Sachiko Isobe, Hideki Hirakawa, Tamotsu Hisamatsu, Yoshihiro Nakano, Masafumi Yagi & Akemi Ohmiya. Genome-wide association study overcomes the genome complexity in autohexaploid chrysanthemum and tags SNP markers onto the flower color genes. Scientific Reports (2019) 9:13947
https://doi.org/10.1038/s41598-019-50028-z
参考図
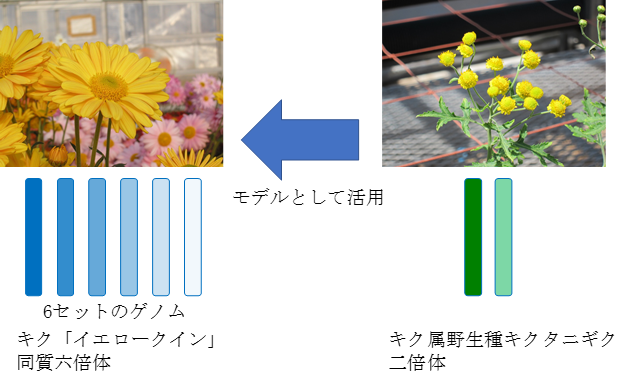 図1 本研究で解析に用いた同質六倍体キク品種「イエロークイン」およびキク近縁野生種で二倍体のキクタニギク
図1 本研究で解析に用いた同質六倍体キク品種「イエロークイン」およびキク近縁野生種で二倍体のキクタニギク
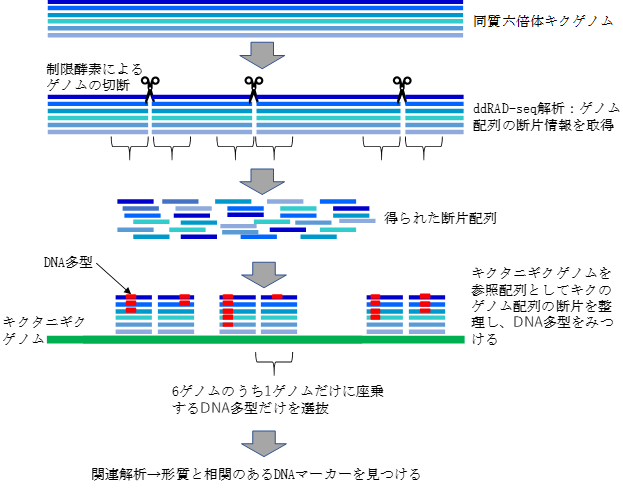 図2 キクにおける効率的DNAマーカー開発手法の概要
図2 キクにおける効率的DNAマーカー開発手法の概要

