開発の社会的背景
タマネギは野菜の中で国内生産量が第3位、国内産出額が約950億円の重要品目です(2020年)。一方で、輸入量が最も多い野菜であり、国内生産量の約1/5を加工業務用として輸入しています。近年のコロナ禍の影響による海外サプライチェーンの不安定化と国内主要産地の不作が重なり、2022年1月から6月までのタマネギの価格は通常時の2倍以上に高騰しました。安定供給のために、実需者からは国産タマネギの増産が求められており、東北・北陸地域を中心に新たな産地形成も進んでいます。産地からは、気象や栽培条件に適し、かつ病気に強い、収量の高い大玉性などの特性を持った品種が求められています。新品種の早期育成には、有用な形質と関連したDNAマーカー(選抜マーカー)を開発し、苗の段階での選抜に利用し、育種を効率化することが必要です。
研究の経緯
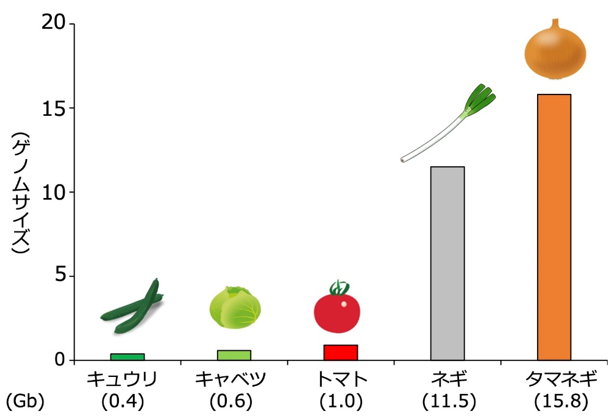
選抜マーカーの開発には、まず染色体全体でのDNA多型を調べ、それらと形質データを照らし合わせて、目的の形質と関連するDNA多型の位置を特定する必要があります。近年は次世代シーケンサーの登場により、大量のDNA情報を安価に解読できるようになったため、染色体全体でのDNA多型の分析が容易になり、多くの野菜品目で選抜マーカーの開発が飛躍的に進みました。一方、タマネギのゲノムサイズは野菜の中でも最大級であり、その大きさはトマトの16倍もあります(図1 )。そのため、他の野菜と同じ手法で染色体全体のDNA多型を分析することは困難であり、選抜マーカーの開発も遅れています。
巨大なゲノムサイズのタマネギでは、効率的なDNA多型の分析のために、発現遺伝子5) が着目されています。生物の細胞では、全てのゲノム情報を利用しているわけではなく、ゲノムに含まれる一部の遺伝子を発現させて、生命の維持活動や葉や根などの器官形成を行います。近年、農研機構が加わった共同研究において、タマネギの葉で発現している約4400個の遺伝子配列情報を取得し、これらの発現遺伝子の染色体上での大まかな並び順を明らかにしました(Fujito et. al., 2021、山口大学他プレスリリース2021年8月20日 URL: http://www.yamaguchi-u.ac.jp/weeklynews/_9039/_9304.html )。
そこで、この情報を利用することにより、染色体上の任意の位置にDNAマーカーが作り易くなり、染色体全体のDNA多型を調べられるDNAマーカーセットを設計できると考えました。また、次世代シーケンサーを活用した技術として、多検体について、多数のDNAマーカーを一度でまとめて分析する手法が開発されました(専門的にはターゲットアンプリコンシーケンス法と呼ばれ、ゲノムの必要な部分のみに焦点を当ててDNA多型を分析する手法)。これらを併せて用いることにより、タマネギにおいて多検体の染色体全体のDNA多型を効率的に分析する手法の確立を目指しました。
研究の内容・意義
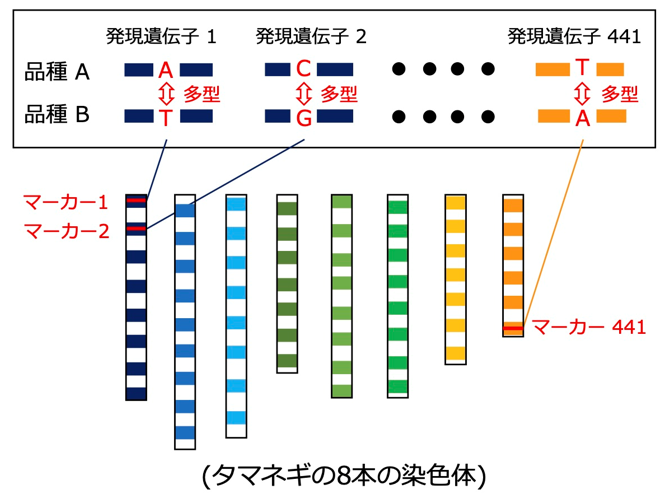
染色体全体のDNA多型を分析できるDNAマーカーセットの設計方法を開発図2 )。まず、解析対象とした2品種(球の大きさや収穫時期が異なる)について、葉の発現遺伝子の配列情報を網羅的に調べて、品種間で配列に違いがあった発現遺伝子を特定しました。次に、先行研究の情報を活用し、配列の違いがあった発現遺伝子の染色体上の位置を推定しました。そして、染色体全体に圴一に分布するように、441個の発現遺伝子を選び、品種間でのDNA多型を分析できるDNAマーカーを作成しました。また、このDNAマーカーセットの設計方法は、解析対象の品種を変えた場合でも有効であり、対象品種に合わせて最適なDNAマーカーセットを効率的に作成できます。
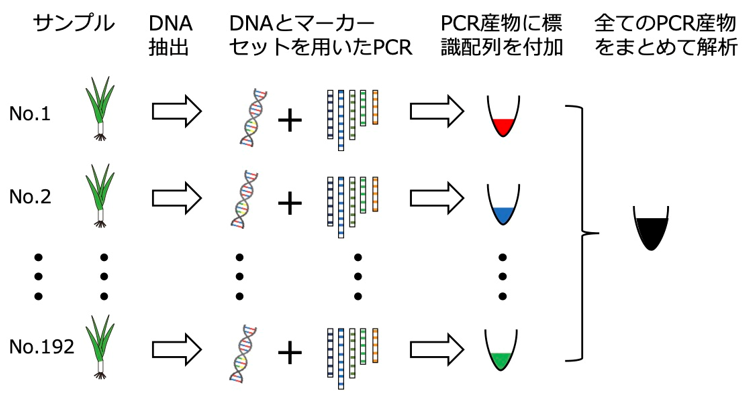
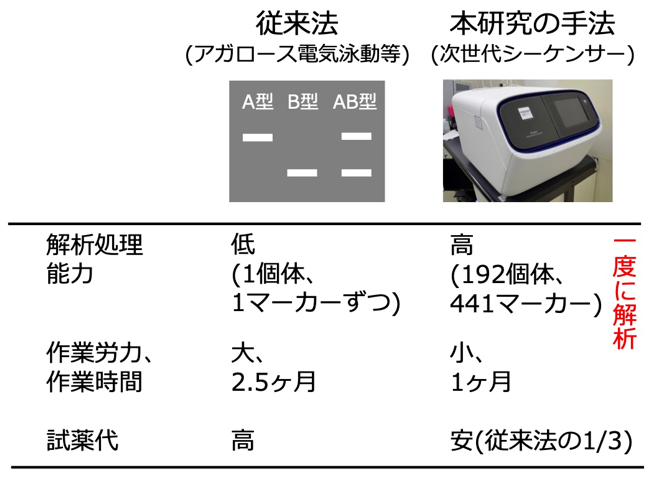
多検体、多数のDNAマーカーを一度に分析する手法の実証図3 )。まず、各個体から抽出したDNAとマーカーセットを用いたPCR6) を行い、各マーカーのDNA多型部分の配列をまとめて増幅しました。次に、各個体のPCR産物に各々の標識配列7) を付加し、その後、全個体のPCR産物を一つにまとめて次世代シーケンサーで解析しました。取得した大量のDNA配列情報を標識配列により個体ごとに分類した後、各マーカーのDNA多型を分析しました。1マーカーずつDNA分析を行う従来法に比べて、分析にかかる作業労力を大幅に減らし、かつ分析にかかる日数を1/2以下、試薬等の分析にかかる費用を1/3以下に減らすことができました(図4 )。得られた個体間のDNA多型と形質データを照らし合わせれば、DNAマーカーセットの中から目的の形質と関連したDNAマーカーを特定でき、選抜マーカーとして利用できるようになります。
今後の予定・期待
今後は本技術の活用により、病気に強い、収量の高い大玉性など各産地や実需者ニーズの高い形質の選抜マーカーの開発を進めていく予定です。これらの選抜マーカーの利用は新品種の早期育成に繋がり、国内生産力強化や安定供給による価格安定に貢献することが期待できます。
用語の解説
ゲノム:
生物の形態を決め、その生存に必要な最小限の染色体セット、またはDNA全体のことをいいます。[ポイントへ戻る]
DNAマーカー:
ゲノム上の特定の位置のDNA多型(塩基配列の違い)を検出できるものを指します。[概要へ戻る]
選抜マーカー:
特定の形質と関連したDNAマーカーを指します。品種育成では、既存の品種に、病気に強い、収量が高いといった、有用な形質を付与することで、新しい品種が育成されます。病気の検定や収穫物の調査は非常に手間がかかりますが、選抜マーカーは有用な形質をもつ個体をDNA多型によって苗の段階で判別できるため、品種育成の効率化に役立ちます。[概要へ戻る]
次世代シーケンサー:
供試した生物種のゲノム全体など、一度に大量のDNA配列を解読できるDNAシーケンサーの総称です。[概要へ戻る]
発現遺伝子:
葉や根などの各器官の細胞内で働いている遺伝子を指します。植物体を構成する細胞は全て同じゲノムを持っていますが、全てを利用しているわけではなく、ゲノムに含まれる一部の遺伝子だけを働かせています。各器官の細胞ごとに発現遺伝子が違うため、それぞれに異なる特徴や機能が生じます。[研究の経緯へ戻る]
PCR:
ポリメラーゼ連鎖反応といい、生物の遺伝情報であるDNAを増幅させる方法を指します。検体から抽出されるのは微量のDNAですが、PCRで特定のDNA配列部分を増やすことで、塩基配列情報を調べることができます。[研究の内容・意義へ戻る]
標識配列:
各個体を識別するための塩基配列です。各個体のPCR産物(各DNAマーカーのDNA多型部分を増幅した塩基配列)に対し、個体ごとに異なる塩基配列を標識として付加しておきます。全個体のPCR産物をまとめて解析した際に、この標識配列を参照することで個体ごとに配列情報を識別できます。[研究の内容・意義へ戻る]
発表論文
Daisuke Sekine, Satoshi Oku, Tsukasa Nunome, Hideki Hirakawa, Mai Tsujimura, Toru Terachi, Atsushi Toyoda, Masayoshi Shigyo, Shusei Sato, and Hikaru Tsukazaki (2022) Development of a genome-wide marker design workflow for onions and its application in target amplicon sequencing-based genotyping. DNA Research. DOI: https://doi.org/10.1093/dnares/dsac020
引用論文
Satoshi Fujito, Turgut Yigit Akyol, Takuya Mukae, Tadayuki Wako, Ken-ichiro Yamashita, Hikaru Tsukazaki, Hideki Hirakawa, Keisuke Tanaka, Yoko Mine, Shusei Sato, Masayoshi Shigyo (2021) Construction of a high-density linkage map and graphical representation of the arrangement of transcriptome-based unigene markers on the chromosomes of onion, Allium cepa L. BMC Genomics. DOI: https://doi.org/10.1186/s12864-021-07803-y
参考図
図1 野菜品目におけるゲノムサイズの違い
図2 染色体全体に配置されたDNAマーカーセットの作成
図3 多検体、多数のDNAマーカーをまとめて分析する手法
図4 従来法と本研究でのDNA分析法の比較