DNA配列断片(k-mer)の照合による外来性DNAの検出方法
要約
組換え体やゲノム編集個体の全ゲノムを解読した大量塩基配列データを、ベクターなどの外来性DNAと効率よく比較照合し、検出できる。ゲノム編集等の社会実装の際に求められるヌルセグレガント(外来性DNAを含まない遺伝的分離個体)の証明を高速かつ高精度に行うために有効である。
- キーワード:ヌルセグレガント、ゲノム編集、新しい育種技術(NBT)、バイオインフォマティクス
- 担当:高度解析センター・ゲノム情報大規模解析チーム
- 代表連絡先:
- 分類:研究成果情報
背景・ねらい
ゲノム編集に代表される新しい育種技術(NBT)で得られた農産品に関しては、最終産物に外来性DNAが不存在(ヌルセグレガント)であれば遺伝子組換えとしては扱わないとされているが、ヌルセグレガントの証明は作出した側が行わなければならない。これはPCRやサザンブロット法などの従来技術でも可能ではあるが精度に問題があり、新たな高精度検出のための技術の開発が求められている。
そこで、本研究では大規模高速ゲノム塩基配列解読と、そのバイオインフォマティクス解析により、短い外来性DNA断片を効率よく検出する方法を検討する。
成果の内容・特徴
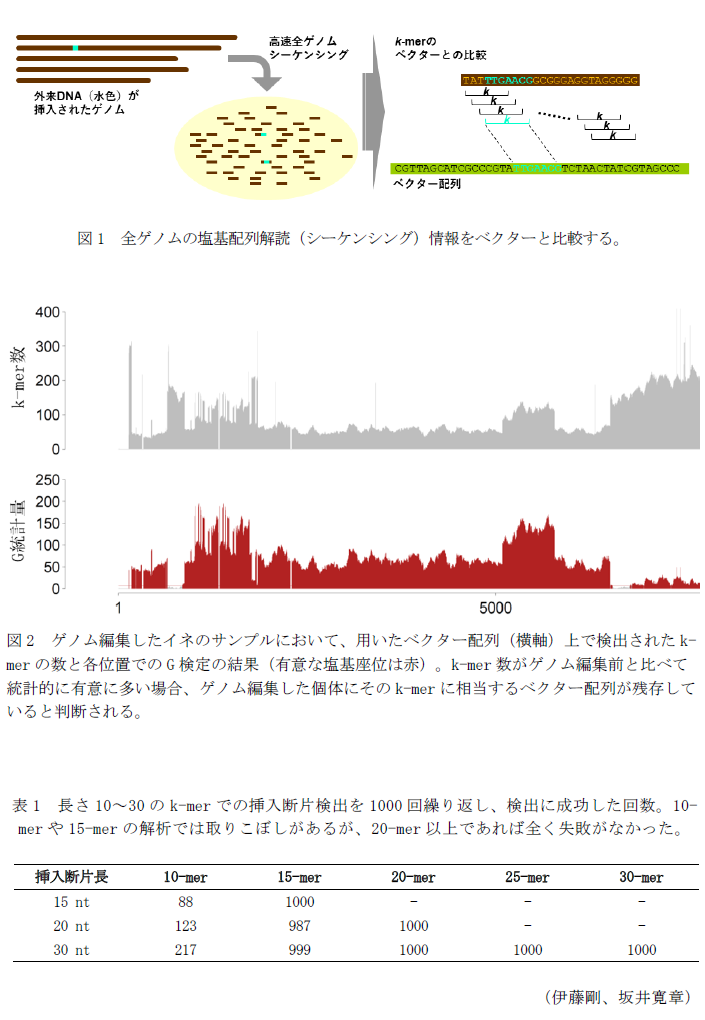
- 本方法は、NBT実行前後の個体から得られた全ゲノム塩基配列を用い、挿入が想定される外来性DNAとの網羅的比較を行うことで検出を行う(図1)。外来性DNAが残存している場合、断片化したDNA配列(k-mer)が野生型より変異体に多く存在し、統計検定によって検出される。
- シミュレーションにより、20塩基程度の断片でも検出されることが確認できる(表1)。このように、k-merの照合による外来性DNA検出は、非常に短い残存断片の高感度検出に対して有効である。
- イネでの実データで、本方法での検出が確認できる(図2)。また、ゲノムが非常に巨大で多倍体であるパンコムギでも同様の有効性が確認できており、多様な生物種に適用が可能である。
成果の活用面・留意点
- 発表論文では20塩基までの検出が可能としたが、これは規制対象に20塩基までの検出を行うべきと主張するものではない。あくまで、一定条件下で技術的限界を検討した結果である。
- DNAシーケンシングのライブラリ構築のための試薬に微量のベクター配列が混入している場合があることが分かっている。これが偽陽性の原因となることがあるので、野生型のゲノム塩基配列解読の段階で想定外のk-mer検出がないかどうか、注意を払う必要がある。
具体的データ
その他
- 予算区分:交付金、その他外部資金(SIP)
- 研究期間:2014~2020年度
- 研究担当者:
伊藤剛、小貫律子、津田麻衣(筑波大)、大嶋雅夫(筑波大)、遠藤真咲、坂井寛章、田中剛、大澤良(筑波大)、田部井豊 - 発表論文等:Itoh T. et al. (2020) Sci. Rep. 10(1):4914
