日本産高級マスクメロンの全ゲノム情報
要約
日本産高級マスクメロンの標準品種「アールスフェボリット春系3号」の全ゲノム情報を解読し、遺伝子情報をデータベース化して公開する。さらに複数のメロン系統のゲノム情報を解読して比較し、レトロトランスポゾンが系統間のゲノムの多様化に寄与した可能性を提唱する。
- キーワード:メロン、長鎖DNAシークエンス、参照ゲノム配列、品種間差
- 担当:高度解析センター・ゲノム情報大規模解析チーム
- 代表連絡先:
- 分類:研究成果情報
背景・ねらい
近年、我が国が誇る高級マスクメロンは海外でも需要が高まっているが、さらに有用な農業形質の付与によるさらなる品質向上と栽培コスト削減が求められている。ゲノム配列と遺伝子情報は、育種や品種デザインにおいてもっとも基礎的かつ必要不可欠な知見であり、全ゲノム情報の解読とデータベースサイトの開発は、国内の育種家や研究者に有用な知見をもたらす。今回、農研機構・野菜花き部門、筑波大と共同して日本産高級マスクメロン標準品種「アールスフェボリット春系3号」の全ゲノム配列情報を解読し、遺伝子情報をMelonet-DBにてデータベース化する。本研究の成果は、国内のメロン品種育成に活用されることが期待される。
成果の内容・特徴
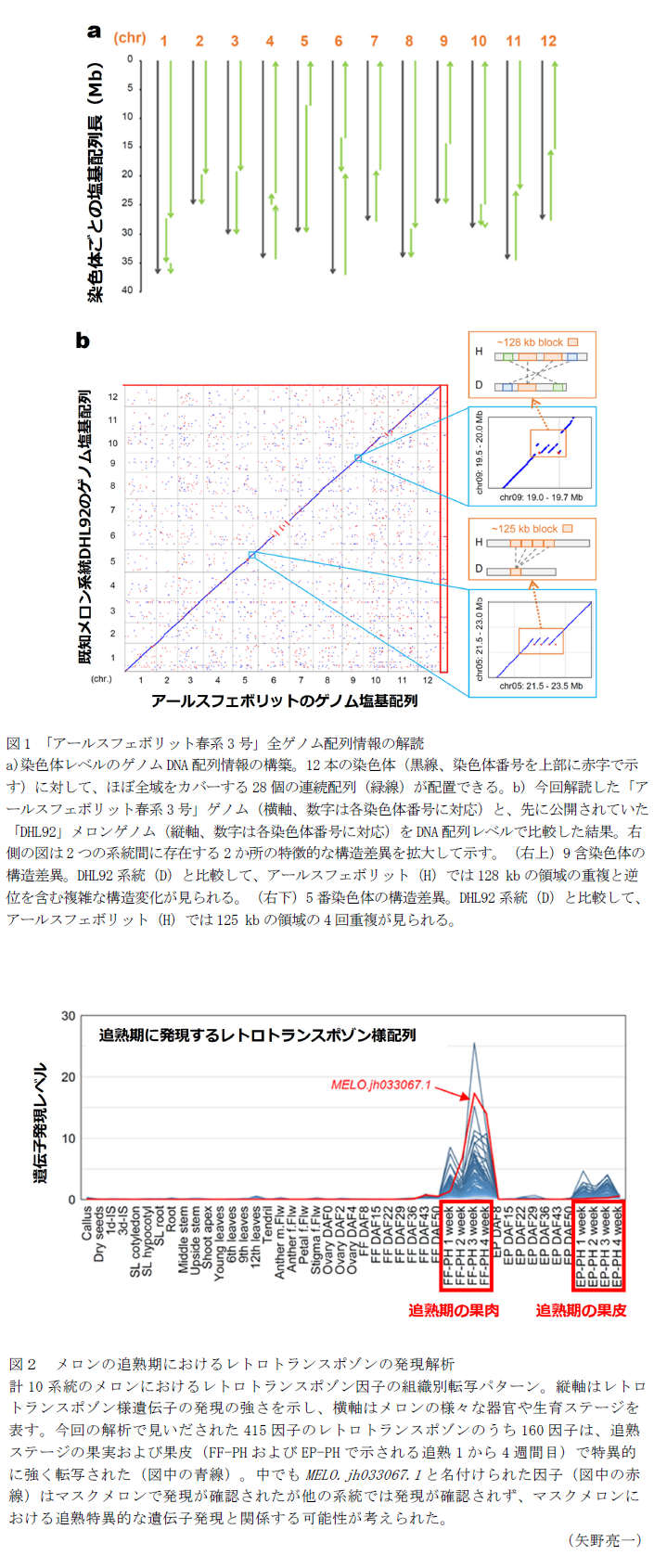
- 従来よりも長いゲノム塩基配列解読が可能な技術(Oxford Nanopore Technology)とを用いることで、「アールスフェボリット春系3号」メロンの12本の染色体ゲノム配列(全長378 Mb)が高精度で解読される(図1)。
- 遺伝子領域を33,829カ所特定し、それらの情報を組織別の遺伝子発現マップと合わせてデータベース化する。また、遺伝子共発現ネットワークを視覚的に解析するための新しいツールを開発して公開する。
- 先に公開されていたスペインのメロン「DHL92」のゲノム情報と比較し、遺伝子コピー数の違いや遺伝子の在不在多型が明らかにされる。
- 「アールスフェボリット春系3号」以外の7つのメロン栽培品種と野生種についても、コンティグ(染色体上で隣り合うDNA配列断片をつなぎ合わせた連続配列)レベルのゲノム配列情報を解読する。系統間にはレトロトランスポゾンの在不在多型が数多く存在することが分かり、それらが重要農業形質に係る遺伝子の新しい発現機能獲得や喪失に係わる可能性が提唱される(図2)。
成果の活用面・留意点
- 「アールスフェボリット春系3号」メロンの参照ゲノム配列情報および遺伝子配列情報は、農研機構のデータベースであるMelonet-DBから取得でき、相同性検索やゲノムブラウザによる閲覧が可能である。また、組織別の遺伝子発現ネットワークなども視覚的に解析することが可能である。今後は、野菜花き部門などと共同して、より大規模に国内品種のゲノム塩基配列データを拡充し、民間企業に提供することを計画している。
- 「アールスフェボリット春系3号」の全ゲノム配列を解読しデータベース化したことにより、香りや食味のみならず、耐病性など様々な農業形質に対して、メロンの品種改良に貢献することが期待される。うどんこ・べと病などのウリ科重要病害の他、近年、他国より侵入してきたウイルス病に対する抵抗性付与育種に活用することが期待される。
具体的データ
その他
- 予算区分:交付金、競争的資金(科研費、戦略的創造研究)
- 研究期間:2018~2020年度
- 研究担当者:矢野亮一、川頭洋一、江面浩(筑波大)、有泉亨(筑波大)、野中聡子(筑波大)
- 発表論文等:Yano R. et al.(2020)Communications Biology 3:432
