植物病原微生物の分類・同定支援システム
要約
農業生物資源ジーンバンクが所蔵する国内産の微生物と、ユーザーの手持ちの微生物の間で遺伝子配列の相同性検索を行い、異同(同種または近縁種等)を推定するシステムである。主として日本産植物病原微生物を効率的に分類・同定する上で有効なツールとなる。
- キーワード:データベース、植物病原微生物、遺伝子相同性検索、分子系統解析
- 担当:遺伝資源センター・微生物分類評価チーム
- 代表連絡先:
- 分類:研究成果情報
背景・ねらい
DDBJ/EMBL/GenBankのデータベースを用いて塩基配列の相同性検索を行うことにより、微生物を分類・同定することが行われている。ただし、その表示学名はデータ登録者の申請情報に由来するため、同定精度は登録情報の質に左右されるという問題がある。そこで、主に日本国内から収集し、農業生物資源ジーンバンクで保存・品質管理している微生物株の配列情報をデータベース化した上で、ユーザーの手持ちの微生物遺伝子情報との間で相同性検索・分子系統解析が実行できるシステムを構築し、日本産植物病原微生物を分類・同定するための支援ツールとして公開する。
成果の内容・特徴
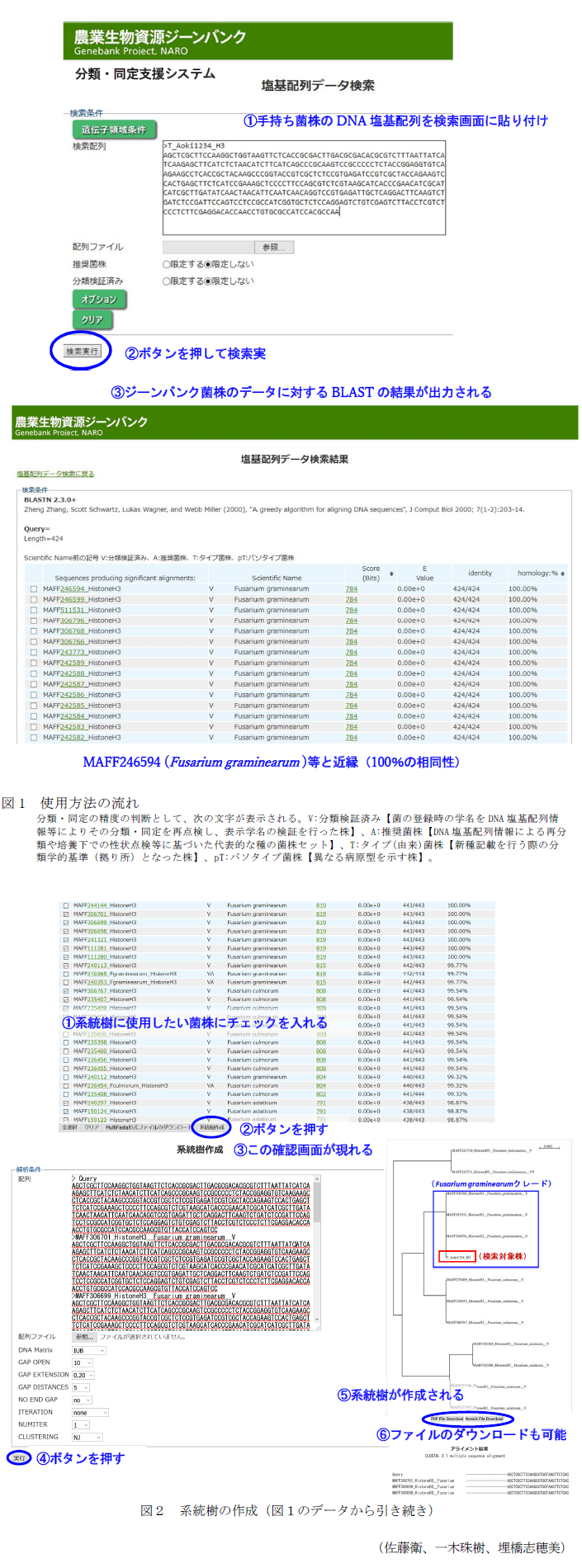
- 本システムは、農業生物資源ジーンバンクの遺伝資源データベース内に設けられたウェブページ(https://www.gene.affrc.go.jp/?db_hom)が入口となる。検索(図1)には、①[遺伝子領域条件]の検索配列欄に手持ちの微生物の分類同定用バーコード遺伝子のDNA/RNA塩基配列を貼り付け、②[検索実行]ボタンを押すと、③ジーンバンク所蔵の微生物(約30,000株の公開株)のデータへ相同性検索された結果が出力される。
- ジーンバンクが管理している微生物との近縁度をもとに、手持ちの微生物を分類し同定するだけでなく、必要に応じて、相同性の高いジーンバンク所蔵微生物を入手することができる。
- ジーンバンクが所蔵する微生物には、「V:分類検証済み、A:推奨菌株、T:タイプ(由来)菌株」等の表示があり、分類・同定の精度を判断できる。
- 検索に用いた手持ち微生物の近縁度の結果を、系統樹として表示することも可能である(図2)。
- 収録されている代表的なバーコード遺伝子としては、糸状菌ではrDNA ITS、TEF1、HIS3、cox1、細菌では16S rRNA、gyrB、rpoD、egl、ウイルスでは外被タンパク質領域等がある。
成果の活用面・留意点
- 日本産植物病原微生物を中心に整備していることから、基礎研究分野における分類研究だけでなく、公設試験研究機関や植物防疫所等での病害の診断にも活用できる。
- サツマイモ基腐病の病原体検出技術の開発において、近縁種との識別が可能な特異的プライマーの設計に本システムが活用されている。
具体的データ
その他
- 予算区分:交付金
- 研究期間:2016~2020年度
- 研究担当者:
青木孝之、一木珠樹、埋橋志穂美、鬼頭英樹、佐藤衛、澤田宏之、竹谷勝、月居佳史、永井利郎、前田美紀、山﨑福容 - 発表論文等:
- Uehara-Ichiki, T. et al. (2020) J. Gen. Pl. Pathol. 86:310-315.
- 澤田宏之ら(2019)植物防疫 73:167-174.
- 埋橋志穂美 (2018)日本微生物資源学会誌 34:13-20.
- 分類・同定支援システム (2021)
