豚増殖性腸炎を引き起こしたローソニア菌の国内初となるゲノムドラフト解析
要約
国内で増殖性腸炎を発症した豚の腸管から抽出した核酸を用い、ローソニア菌の全ゲノムドラフト解析を行った結果から、国内の3株は米国で報告された豚由来株2株に比べ遺伝的多様性が大きく、宿主適応に関係するとされるゲノム領域を欠くことが分かる。
- キーワード:ローソニア菌、増殖性腸炎、全ゲノム、ドラフト解析、宿主適応
- 担当:動物衛生研究部門・細菌・寄生虫研究領域・細胞内寄生菌ユニット
- 代表連絡先:電話029-838-7791
- 分類:研究成果情報
背景・ねらい
ローソニア菌(Lawsonia intracellularis)はグラム陰性の偏性細胞内寄生細菌であり、世界中に分布している。本菌は宿主域が広く種々の動物に感染するが、特に豚や馬に感染すると腸管の上皮細胞内で増殖し、難治性下痢症である増殖性腸炎を引き起こす。増殖性腸炎を起こした腸管は本来の栄養吸収能が低下するため、飼料効率の悪化や増体量の減少を招く。本菌の国内農場浸潤率は95%以上との報告もあることから、産業界に与える経済的損失は甚大である。ローソニア菌の人工培地を用いた培養は未だ成功していない。このため感染経路や病原因子などの詳細はもちろん、遺伝子解析もほとんどなされておらず、ゲノム解読もこれまで米国での3株(豚由来2株、馬由来1株)のみにとどまっている。
本研究では国内の3県で増殖性腸炎と診断された豚の腸管から核酸を抽出し、国内初となるローソニア菌のゲノムドラフト解析を行って、米国の豚由来株と比較することで、病原因子等の不明な点について新たな知見を得ることを目的とする。
成果の内容・特徴
- 全ゲノム配列が決定されている豚由来株(PHE/MN1-00株)を参照配列とし、得られた国内豚由来株のゲノムショート配列をマッピングすることにより、宿主である豚や夾雑菌のゲノム配列を除いている。これにより、ローソニア菌を対象としたゲノム解析が可能となる。
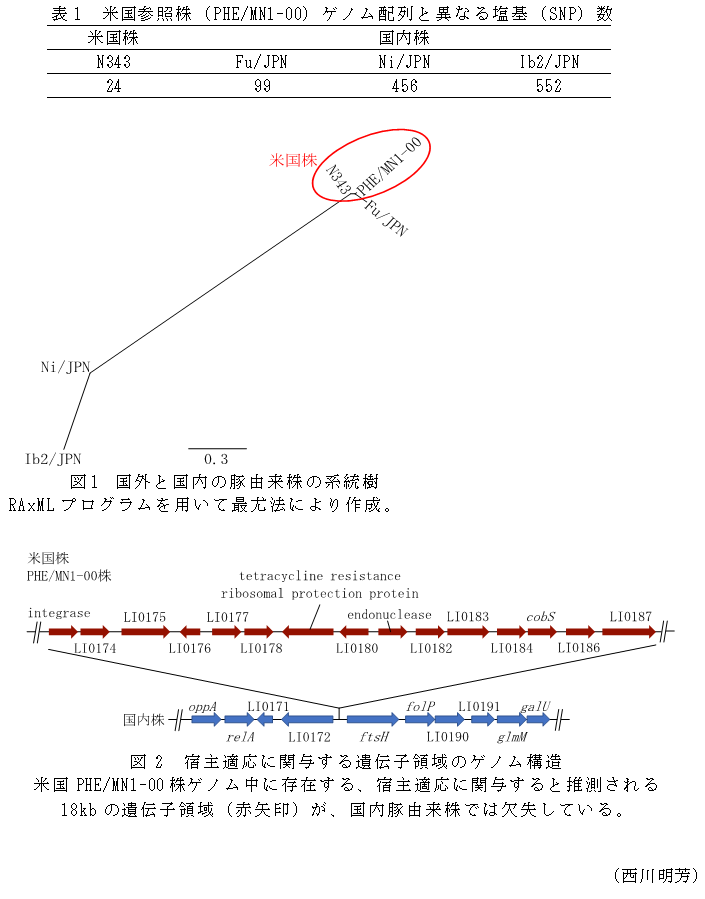
- PHE/MN1-00株参照配列と米国豚由来株(N343株)のゲノム配列とを比較すると一塩基多型(SNPs)は24個であるのに対し、得られた国内株Fu/JPN、Ni/JPN、Ib2/JPN株を比較するとそれぞれSNPsは99、456、552個である。Fu/JPN株は米国株と近縁であるが他の国内株2株は近縁でなく、国内株は米国株に比べて遺伝的多様性が大きいことが分かる(表1、図1)。
- 米国PHE/MN1-00株のゲノムに中に存在する、豚への宿主適応に関与すると推測される18kbの遺伝子領域は国内豚由来株では欠失している(図2)。
成果の活用面・留意点
- 3株(Fu/JPN、Ni/JPN、Ib2/JPN)の決定したゲノム配列はDNA Database of Japan (DDBJ)のデータベースに登録及び公開済みである(アクセッション番号:QNHO00000000[Fu/JPN]、QNHN00000000[Ni/JPN]、QNHM00000000[Ib2/JPN])。
- 公開済の配列データにはインターネットを通じて誰でもアクセスできるため、世界中で本研究成果を利用した増殖性腸炎研究の進展が期待できる。
具体的データ
その他
- 予算区分:交付金
- 研究期間:2018年度
- 研究担当者:西川明芳、小川洋介、江口正浩、Anura Rambukkana(エジンバラ大学)、下地善弘
- 発表論文等:Nishikawa S. et al. (2018) Microbiol. Resour. Announc. 7:e01021-18
