カイコの高精度参照ゲノム配列情報
要約
第三世代シーケンサーを用いて新規に構築したカイコのゲノム配列情報である。既存のカイコゲノム配列情報と比較して、より正確かつ網羅性が高い高精度のゲノム配列情報であり、カイコによる有用物質生産、重要害虫の新規防除法開発などの研究の加速化への貢献が期待される。
- キーワード:カイコ、第三世代シーケンサー、参照ゲノム配列、解毒分解関連遺伝子
- 担当:生物機能利用研究部門・遺伝子利用基盤研究領域・先進昆虫ゲノム改変ユニット
- 代表連絡先:電話 029-838-7007
- 分類:研究成果情報
背景・ねらい
カイコゲノム情報は日中の共同研究によって2008年に解読され、カイコおよび重要害虫などに関する様々な研究開発において重要な役割を果たしている。しかし、既存のカイコゲノム情報には配列情報が未決定または不正確と思われる領域が局所的に存在しており、更なる高精度化が求められている。そこで、東京大学、農研機構、遺伝学研究所による共同研究により、第三世代シーケンサーを活用してカイコゲノム情報の更なる高精度化を実現し、その有用性を明らかにする。これにより、カイコによる有用物質生産、重要害虫の新規防除法開発などの研究の加速化に貢献する。
成果の内容・特徴
- 新規に構築した参照ゲノム配列は、カイコp50T系統の雄個体における全染色体(28対)の塩基配列を第三世代シーケンサー(PacBioRS IIシステム)を用いて解読したものである。
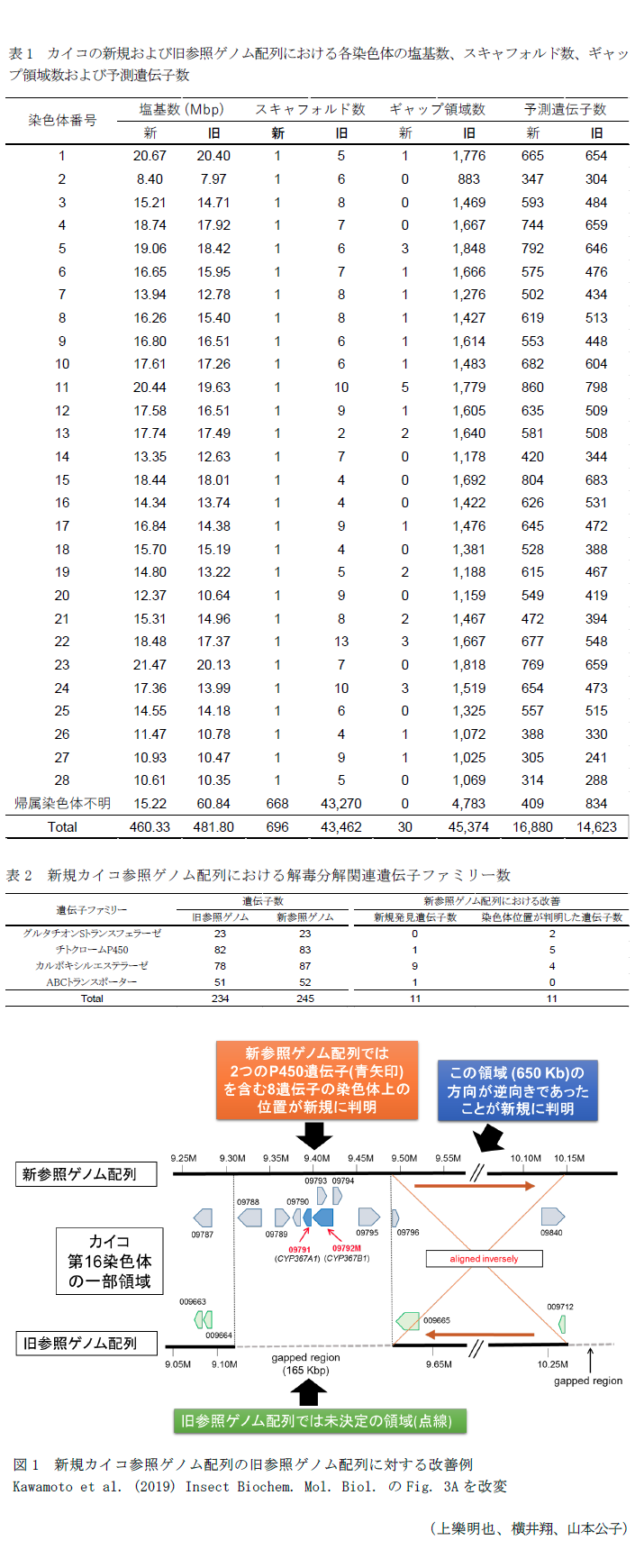
- 新規参照ゲノム配列の全長は460.3Mbである。未決定領域は30箇所のみで、各染色体についてほぼ完全な一本の塩基配列を決定しており、16,880個の遺伝子 が予測されている(表1)。
- 農薬の解毒分解などに関連する4種の遺伝子ファミリーについて、遺伝子配列情報を手作業で詳細に検証し、より正確な遺伝子配列情報を決定している(表2)。
- 新規参照ゲノム配列では、既存の参照ゲノム配列では未決定の領域や配列情報が不正確な領域が多数改善されている(図1)。
成果の活用面・留意点
- 新規参照ゲノム配列情報および遺伝子配列情報は、東京大学のSilkBaseおよび農研機構のKAIKObaseから取得でき、相同性検索やゲノムブラウザによる閲覧が可能である。
- 新規参照ゲノム配列情報は、カイコにおける有用物質生産などの有用形質に関連する遺伝子マーカーの開発、カイコと同じチョウ目に属する重要害虫の薬剤抵抗性原因遺伝子の同定およびこれを用いた抵抗性モニタリング法の開発などに活用できる。
具体的データ
その他
- 予算区分:交付金
- 研究期間:2017~2018年度
- 研究担当者:
上樂明也、横井翔、山本公子、川本宗孝(東京大)、豊田敦(遺伝研)、水口洋平(遺伝研)、勝間進(東京大)、藤山秋佐夫 (遺伝研)、木内隆(東京大)、嶋田透(東京大) - 発表論文等:Kawamoto M. et al. (2019) Insect Biochem. Mol. Biol. 107:53-62
