FnCas12a標的配列長の最適化による高効率ゲノム編集
要約
ゲノム編集酵素の一種であるFnCas12aと複合体を形成し、標的配列の認識を担うcrRNA中に配する標的配列の長さを最適化することにより、FnCas12aを用いたゲノム編集効率を向上させることが可能である。
- キーワード:ゲノム編集、CRISPR、FnCas12a、DNA切断
- 担当:生物機能利用研究部門・遺伝子利用基盤研究領域・先進作物ゲノム改変ユニット
- 代表連絡先:
- 分類:研究成果情報
背景・ねらい
Casタンパク質自体が認識するゲノム配列はPAM配列と呼ばれ、CRISPR/Cas9システムによるゲノム編集において標的配列の制限要因となっている。現在最も広く利用されているSpCas9はNGGをPAM配列として認識し、平滑末端型のDNA二重鎖切断を導入するため、GCリッチな領域のゲノム編集を得意とする。一方、FnCas12aはTTNをPAM配列として認識し、突出末端型のDNA二重鎖切断を導入することから、ATリッチな領域のゲノム編集や、突出末端の形成が鍵となる、鋳型DNAをコピーするタイプのゲノム編集、「標的組換え」に有利であると考えられるが、一般的にFnCas12aのDNA切断効率はSpCas9に比べて低い。変異導入には、標的配列の認識能も影響することから、本研究では、FnCas12aと複合体を形成し、標的配列の認識を担うcrRNAの鎖長を最適化し、FnCas12aを用いたゲノム編集効率の向上を目指す。
成果の内容・特徴
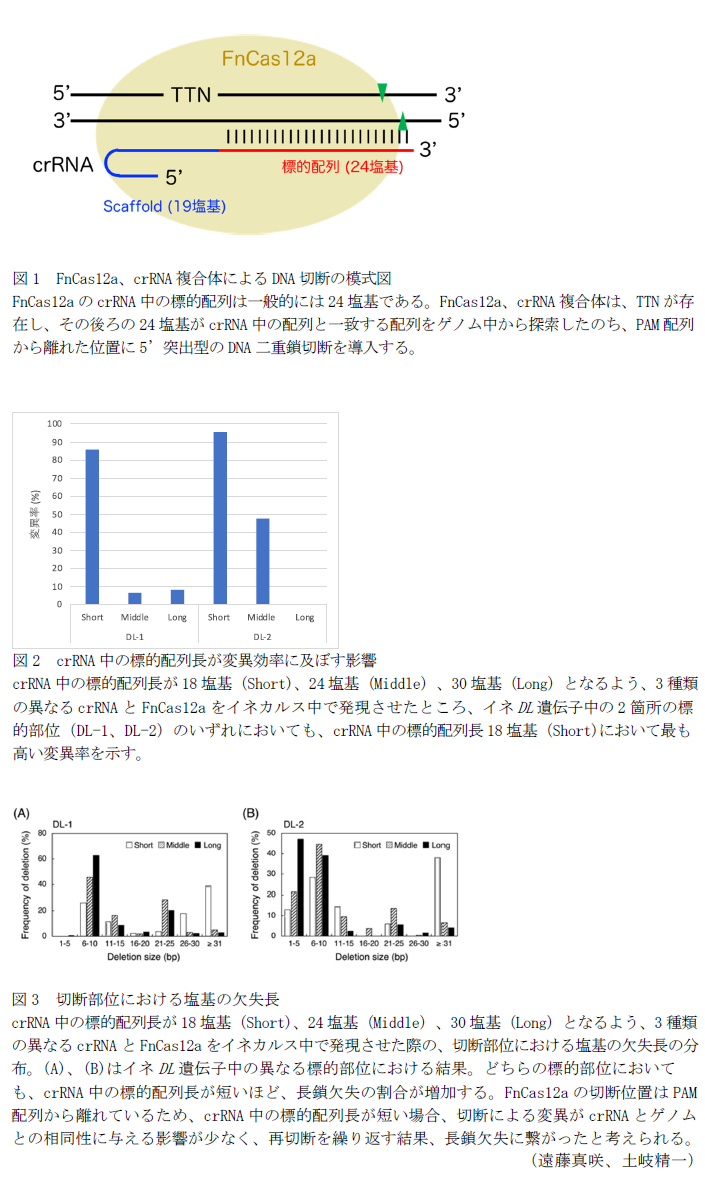
- FnCas12aを用いたゲノム編集系においては、crRNA中に配する標的配列は一般的に24塩基であるが(図1)、イネDL遺伝子中の2箇所の標的サイトにおいては、crRNA中に配する標的配列を18塩基に変更することにより、変異効率が向上する(図2)。
- 上記実験において18塩基の標的配列を含むcrRNAを用いた場合、切断部位の塩基欠失長が24塩基の標的配列の場合よりも長くなる傾向が見られる(図3)。
普及のための参考情報
- crRNA中に配する標的配列が18塩基の場合、24塩基の場合と比べて、変異率が高く、塩基欠損長が長い傾向はあったが、逆に通常より長い30塩基にすることで変異率が上昇する場合もあり、標的配列により最適な鎖長は異なると考えられ、個別に検討する必要がある。
- FnCas12aは切断部位がPAM配列から離れていることから、変異が導入されても標的配列に変化がないためDNA切断を繰り返し生じ、結果的に長い欠失が起きる可能性がある。連続したDNA切断は変異の多様性拡大に有利である。また、標的組換えと呼ばれるゲノム編集おいては、DNAの突出末端の存在が重要となるため、本方法との併用によって、効率向上の可能性がある。
具体的データ
その他
- 予算区分:その他外部資金(SIP)
- 研究期間:2016~2020年度
- 研究担当者:根岸克弥、三上雅史、土岐精一、遠藤真咲
- 発表論文等:Negishi K. et al.(2020)Frontiers in Genome Editing doi: 10.3389/fgeed.2020.608563
