ダイズのサポニン組成を制御する遺伝子群
要約
ダイズのサポニン組成には4種類の自然変異体が存在する。同定した各原因遺伝子が変異した国産優良品種「エンレイ」の誘発突然変異体は、自然変異体と同様のサポニン組成を示す。遺伝子情報ならびに変異体を利用することにより、ダイズの食味や健康機能性の効率的な育種的改良が可能である。
- キーワード:大豆、遺伝資源、突然変異体、食味、機能性
- 担当:次世代作物開発研究センター・基盤研究領域
- 代表連絡先:電話029-838-7930
- 分類:研究成果情報
背景・ねらい
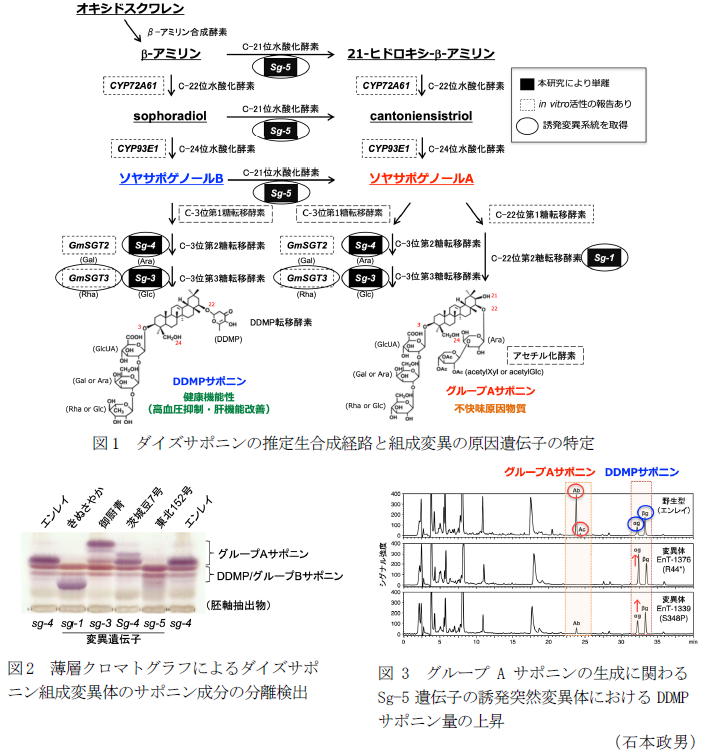
ダイズサポニン(ソヤサポニン)はトリテルペノイドサポニンに分類され、アグリコン(非糖部)に糖が付加した配糖体の総称である。マメ科植物に広く分布し、根から環境中に放出される事が報告されているが、植物における生理機能の詳細はわかっていない。ダイズでは種子や根に高濃度に含まれている。ダイズサポニンはグループAサポニンとDDMPサポニンに大別される。グループAサポニンは不快味原因物質であるが、DDMPサポニンとその分解産物(グループB,Eサポニン)は健康機能性を示す(「大豆と日本人の健康」渡邊昌監修)。ダイズのサポニン組成に関しては、遺伝資源の探索からsg-1、sg-3、Sg-4、sg-5の4種類の組成変異遺伝子が見つかっている(図1、図2)。なかでも、sg-1とsg-5はグループAサポニンの化学構造と有無を制御することから品種育成への利用が進められている。そこで、本研究では、食味や健康機能性に優れたダイズ品種の効率的な育成のために、これら4種類の自然変異体の原因遺伝子を単離するとともに、育種素材として各原因遺伝子が変異した国産優良品種「エンレイ」の誘発突然変異体を選抜する。
成果の内容・特徴
- 連鎖解析に基づき、sg-1、sg-3、Sg-4、sg-5の4種類の組成変異の原因遺伝子を単離した。
- グループAサポニンのC-22位の第2糖を付加するSg-1座には、キシロースを付加するSg-1a(UGT73F4)、グルコースを付加するSg-1b(UGT73F2)、糖を付加できないsg-10の3つの対立遺伝子が存在する。このうち、sg-10の利用によりアセチル化された第2糖が付加しないため、食味の改善が期待できる(図1)。
- C-3位末端のグルコースの付加に関わるSg-3座は糖転移酵素(UGT91H9)をコードしており、同じ位置にラムノースを付加するGmSGT3(UGT91H4)との二重変異によりC-3位の末端糖が付加しないサポニンを蓄積する(図1)。
- C-3位第2糖にアラビノースを付加するSg-4座は糖転移酵素(UGT73P10)をコードする(図1)。
- グループAサポニンの生成に関わるSg-5座は、C-21位の酸化に関わるCYP72A69をコードしている。この遺伝子の機能欠損型変異sg-5によりDDMPサポニンが増加することから、食味や健康機能性の改良が期待できる(図1、図3)。
- 各遺伝子について国産優良品種「エンレイ」の誘発突然変異体を得て、それらのダイズサポニン組成が自然変異体と同様であることを確認している。
成果の活用面・留意点
- 食味や機能性に関わるサポニン組成の育種的改良に利用できる。
- 利用する自然あるいは誘発変異体によって、アリル型識別用のDNAマーカーセットを使い分ける必要がある。
- 誘発変異体には目的遺伝子座以外にも塩基置換変異が誘発されているため、遺伝資源同様、戻し交配等によって不良な領域(変異)を取り除く必要がある。
具体的データ
その他
- 予算区分:交付金、委託プロ(次世代ゲノム)、競争的資金(イノベ創出強化)
- 研究期間:2010~2018年度
- 研究担当者:石本政男、矢野亮一、髙木恭子、高田吉丈、佐山貴司、加賀秋人、穴井豊昭(佐賀大)、小埜栄一郎(サントリーグローバルイノベーションセンター(株))、關光(大阪大)、村中俊哉(大阪大)、塚本知玄(岩手大)
- 発表論文等:
- Sayama T. et al. (2012) The Plant Cell 24:2123-2138
- Yano R. et al. (2017) The Plant Journal 89:527-539
- Yano R. et al. (2018) Plant and Cell Physiology 59:797-810
- Takagi K. et al. (2018) Phytochemistry 156:96-105
