次世代シーケンサーによるダイズのゲノムワイドマーカーセットと解析パイプライン
要約
国内外のダイズにおいて次世代型シーケンサーを用いてゲノムワイドに解析可能で、かつ重要形質遺伝子の識別ができるマーカーセットである。本解析パイプラインを用いることで、得られた塩基配列データを自動的に遺伝子型データに変換し、表現型の予測に利用することができる。
- キーワード:ダイズ、ゲノムワイドマーカー、次世代シーケンサー、解析パイプライン
- 担当:次世代作物開発研究センター・畑作物研究領域・畑作物形質評価ユニット
- 代表連絡先:電話 029-838-7135
- 分類:研究成果情報
背景・ねらい
ダイズでは、重要形質遺伝子の塩基配列やゲノムに散在する多数の一塩基多型(SNP)などを次世代型シーケンサーによって解析できる環境が整いつつある。本研究では、重要形質遺伝子を含むゲノムワイドなマーカーを設定し、次世代シーケンサーを用いて解析することで、従来の方法では困難であった重要形質遺伝子の責任変異の検出と、大規模な遺伝子型データの取得を実現するとともに、得られたデータを自動的に処理できる解析基盤を開発し、育種選抜の高精度化とハイスル-プット化を目指す。
成果の内容・特徴
- 国内外のダイズ品種のゲノム情報および遺伝情報に基づき、遺伝子上に3062個、遺伝子間領域に1052個、合計4114個でゲノム全体をカバーするように配置したマーカーセットである。
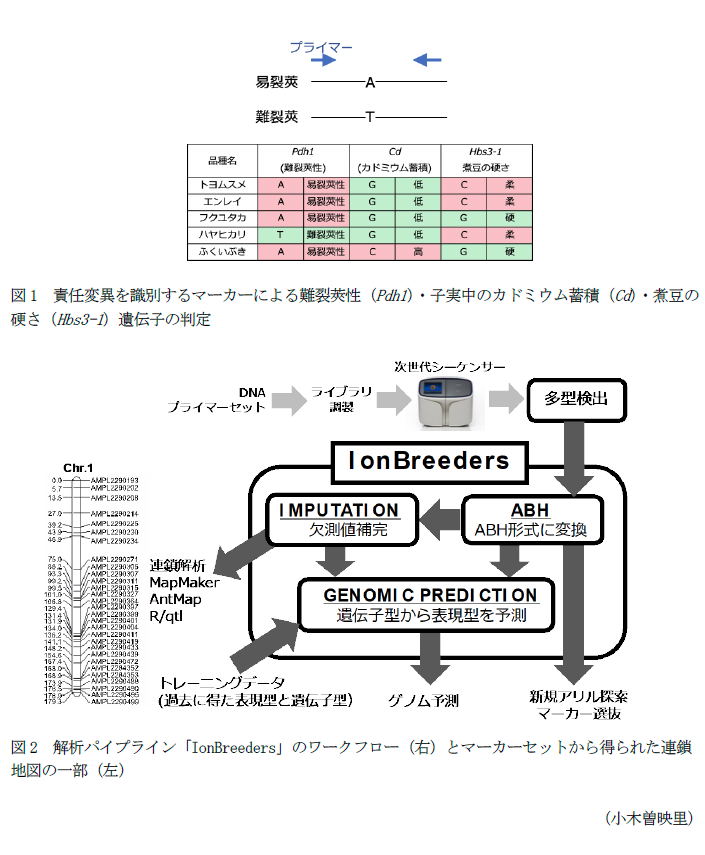
- このマーカーセットには、ダイズの重要形質である難裂莢性(Pdh1)、カドミウム含量(Cd)、リポキシゲナーゼ欠損(L2-loss)、煮豆の硬さ(Hbs3-1)、主要開花関連遺伝子(E1, E2, E3, E4)、葉形(Ln)、シストセンチュウ抵抗性(rhg1a, Rhg4)、伸育性(Dt1)等に関する遺伝子の責任変異を識別できるマーカーを含む(図1)。
- このマーカーセットは自由な組み合わせで、ウルトラマルチプレックスPCR法によって一度に増幅することができ、増幅産物から調整したライブラリを次世代シーケンサーで解読することで、一度に最大768サンプルを解析できる。
- 解析パイプラインは、次世代シーケンサーの塩基配列解読データから、汎用的なマーカー遺伝子型情報への変換、信頼性の低いデータの除去、欠測データの補完など、一連の作業を半自動的に行うことができる(図2)。
- 解析パイプラインには、過去のデータ等、複数のマーカー遺伝子型と表現型のデータセットをトレーニングデータとして取り込むことで、様々なマーカー遺伝子型の個体が取りうる表現型を予測して出力する「ゲノム予測」(genomic prediction)の機能を含む(図2)。
成果の活用面・留意点
- 責任変異をマーカーとして利用したことで、DNA多型情報に基づき高い精度で標的形質を評価できる。
- 既存のマーカーセットの情報を考慮して新たに追加したい責任変異マーカーをデザインすることで、新たなマーカーと既存のマーカーとを一度にまとめて増幅し、解析することが可能である。
- 解析パイプラインはGithubからダウンロードでき、生物種を問わず利用できる。
- 近縁度が高く多型が十分に得られない交配組み合わせでは、マーカーを追加する必要がある。
具体的データ
その他
- 予算区分:交付金、委託プロ(次世代ゲノム)、その他外部資金(27補正「先導プロ」)
- 研究期間:2015~2019年度
- 研究担当者:小木曽映里、田口文緒、矢部志央理、田中剛、加賀秋人、羽鹿牧太、石本政男
- 発表論文等:
- Ogiso-Tanaka E. et al. (2018) J Appl Microb Res. 1:23-49
- Ogiso-Tanaka E. et al. (2019) DNA Res. 26:243-260
- Ogiso-Tanaka E. et al. (2020) Breed. Sci. 70:396-401
- 小木曽 (2019) 職務作成プログラム「IonBreeders」、機構V-03
