カンキツのDNAマーカー開発を支援するミカンゲノムデータベース(MiGD)
要約
ミカンゲノムデータベース(MiGD)は、ウンシュウミカンとカラタチのゲノム配列、10種類のカンキツ及びその近縁種間のDNA多型、約2,700種類のCAPSマーカーの情報が閲覧でき、カンキツの有用遺伝子の特定や、新品種育成過程での優良個体の選抜、品種鑑定を効率化できる。
- キーワード:カンキツ、ゲノム、データベース、品種識別、DNAマーカー
- 担当:果樹茶業研究部門・カンキツ研究領域・カンキツゲノムユニット、高度解析センター
- 代表連絡先:電話 029-838-6436
- 分類:普及成果情報
背景・ねらい
カンキツでは、優れた新品種の育成を効率化するため、優良形質の幼苗選抜に利用できるDNAマーカーの開発や、育成者権保護のためのDNA鑑定技術の開発に必要なDNAマーカーの開発を進めている。DNAマーカーの開発には、クレメンティンやオレンジなど海外で多く栽培されるカンキツの全ゲノム情報を利用しているが、国内の優れた育種素材を最大限に活用して新品種を効率的に育成するためには、国内で広く栽培されている在来種や育種素材の全ゲノム配列を解読し、それらのゲノム配列情報やDNA多型情報などが簡単に閲覧できる操作性の優れたデータべースの開発が必要である。そこで、カンキツの有用遺伝子の特定や、新品種育成過程での優良個体の選抜、品種鑑定のためのDNAマーカーの開発を支援するデータベースを開発する。
成果の内容・特徴
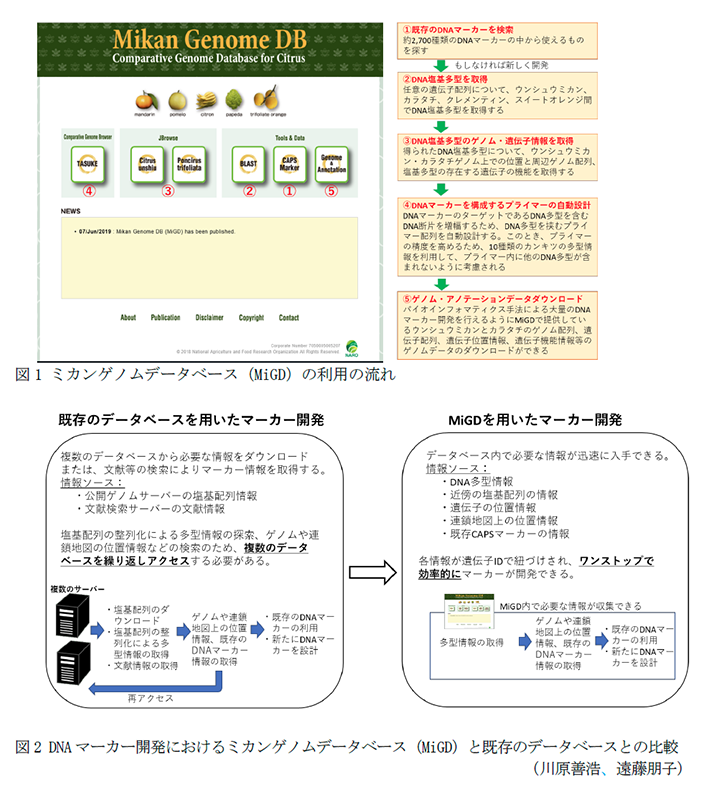
- MiGDは、高精度化されたウンシュウミカンと世界ではじめて解読したカラタチのゲノムアノテーションデータベース、DNA多型データベース、CAPSマーカーのデータベースを遺伝子IDで紐づけた統合型ベースであり、ワンストップで効率的にDNAマーカーを開発できる。
- ゲノムアノテーションデータベースでは、独自にアセンブルしたウンシュウミカンとカラタチの全ゲノム情報をゲノムブラウザ「JBrowse」により、任意の遺伝子の構造情報、配列情報、遺伝子機能情報が検索・閲覧できる。
- 全ゲノムを構成するサイズ、スキャフォールド数および予測遺伝子数は、ウンシュウミカンで、3億5千万塩基対、3,151スキャフォールド、カラタチで41,489遺伝子、2億9千万塩基対、1,313スキャフォールド、34,333個の遺伝子である。
- DNA多型データベースでは、クレメンティンのゲノム配列を参照配列としてウンシュウミカン、オレンジ、ポンカン、ブンタンなど10種類のカンキツ間に存在する一塩基多型(SNP)情報をゲノムブラウザ「TASUKE」により検索・閲覧できる。
- CAPSマーカーデータベースでは、約2,700種類のCAPSマーカーのプライマー配列情報、これまで作成した連鎖地図、クレメンティンやウンシュウミカンの公開全ゲノム配列における位置情報代表的なカンキツ品種のCAPSマーカーの遺伝子型情報などが検索・閲覧できる.
- MiGDは「https://mikan.dna.affrc.go.jp/」のサイトで公開されている。
普及のための参考情報
- 普及対象:農業試験研究機関、大学
- 普及予定地域・普及予定面積・普及台数等:カンキツの育種事業を行う公設試等
- その他:アクセス件数25.2万件(2019年12月9日時点)。公開するデータは随時更新する予定である。
具体的データ
その他
- 予算区分:交付金、委託プロ(新ゲノム育種)
- 研究期間:2014~2019年度
- 研究担当者:遠藤朋子、川原善浩(高度解析センター)、大村三男(静岡大)、寺本由美子((株)情報処理バイオ)、伊藤剛(高度解析センター)、藤井浩、島田武彦
- 発表論文等:Kawahara et al. (2020) Breeding Science 70(2):200-211
