キク栽培品種を利用した網羅的EST解析
要約
キク栽培品種「セイマリン」由来の発現遺伝子配列断片(expressed sequence tag、EST)情報の網羅的解析により構成された213,204個の相同連結配列(コンティグ)情報をキクのゲノム育種研究のための基盤情報として公共データベース上で公開している。
- キーワード:キク、EST、コンティグ、データベース
- 担当:野菜花き研究部門・花き遺伝育種研究領域・遺伝子制御ユニット
- 代表連絡先:電話029-838-6805
- 分類:研究成果情報
背景・ねらい
キクでは主に、交配育種や突然変異育種によって新品種が作出されている。一方、様々な植物種においてDNAマーカー育種が進みつつあり、また近年のゲノム編集技術の急速な進展により、今後はゲノム編集技術を利用した育種も期待されている。これら次世代の育種技術には遺伝子配列情報が不可欠であるが、キクは6倍体かつゲノムサイズが12Gbp以上と大きく、また栽培品種も非常に多いことから、配列情報の整備が進み難くゲノム育種研究のための遺伝子配列情報が不足している。一方で、年々、次世代シークエンスの性能の進展や低価格化が進んでおり、今後はキクにおいてもゲノム育種研究の情報を利用した新品種開発が進展することが期待される。本研究では、キクにおけるゲノム育種研究の発展に資する配列情報を提供することを目的に、発現遺伝子配列断片(expressed sequence tag、EST)情報を網羅的に解読する。
成果の内容・特徴
- キク栽培品種である「セイマリン」の地上部の植物器官を材料に用い、次世代シーケンス技術を利用して得られた高精度配列数は、3,722,677リードであり解読塩基数は約1,702Mbpである(表1)。
- 得られたEST配列のうち、相同配列を連結して整理統合すると、213,204個の相同連結配列(コンティグ)の配列情報が得られる(表1)。得られたコンティグ配列を全ゲノム配列が解読されたシロイヌナズナの遺伝子配列と比較すると、全コンティグの約61%がシロイヌナズナ遺伝子配列と高い相同性を示す。この結果はイネとシロイヌナズナの配列比較解析の結果とほぼ同様であり、これらの約61%の遺伝子は、植物全体に広く共通する遺伝子配列であることが示される。
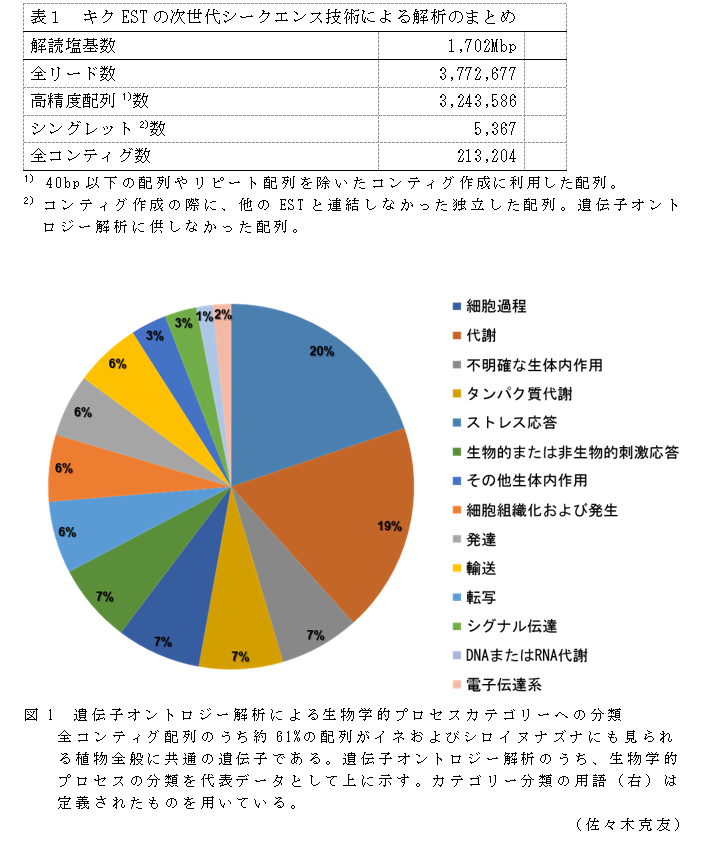
- 遺伝子オントロジー解析を行うと、このEST情報は多くの生物学的プロセスに関係する遺伝子配列を含んでいる(図1)。この生物学的プロセスのうち、得られたEST配列の20%が細胞過程に関する、また19%が代謝に関する遺伝子配列を含んでいる。
成果の活用面・留意点
- EST情報の全ての生データ配列は国際塩基配列データベース(DDBJ/EMBL/GenBank)より登録番号DRR088291-DRR088294として公開されている。全コンティグ配列については、IABW01000001-IABW01213204として個別に公開されている。
- キクの品種改良や品質向上に資する可能性のある遺伝子を本EST情報から簡便に探索でき、遺伝子解析、マーカー開発およびゲノム編集等に有用な情報である。
- 遺伝子オントロジー解析とは、データベース化されている遺伝子関連情報を基に、得られた網羅的遺伝子配列を「生物学的プロセス(生物内で起こる反応・イベント等)」、「細胞の構成要素(細胞内の局在、構成名称等)」および「分子機能(分子および物質の名称等)」の3つのカテゴリーに分けるなど、遺伝子群ごとに解析する手法である。これにより、遺伝子の機能や細胞内局在がある程度推定できる。
具体的データ
その他
- 予算区分:交付金
- 研究期間:2011~2017年度
- 研究担当者:佐々木克友、光田展隆(産総研)、奈島賢児(日本大)、岸本久太郎、片寄裕一、金森裕介、大宮あけみ
- 発表論文等:Sasaki K. et al. (2017) BMC Genomics. 18:683
