キク二倍体近縁野生種キクタニギクの全ゲノムの塩基配列の解読
要約
キク近縁野生種キクタニギク(Chrysanthemum seticuspe)において全ゲノムの89%に相当する27.2億塩基を解読し、推定遺伝子数は71,057である。キクのモデルとして配列および遺伝子情報を利用できる。
- キーワード:キク、ゲノム解読、キク科、遺伝子
- 担当:野菜花き研究部門・花き遺伝育種研究領域・ゲノム遺伝育種ユニット
- 代表連絡先:電話 029-838-6805
- 分類:研究成果情報
背景・ねらい
キクは、わが国で最も生産量の多い花きである。花色、病害抵抗性や開花特性などの重要形質を制御する遺伝機構を解明し、品種改良を加速するためにゲノム情報は極めて有用である。一方、キクは他殖性の同質六倍体であり、遺伝的なヘテロ性が高く、ゲノムサイズが大きいことからゲノム解読は困難である。キクタニギクは、東北から九州にかけて自生するキク属の二倍体近縁野生種である。キクと同様の生育開花反応を示し、キクに比べてゲノム構造が単純なため、キクのモデル植物として研究に利用されている。そこで、本研究では栽培ギクのゲノム解析基盤として、キクタニギクの全ゲノムを解読する。
成果の内容・特徴
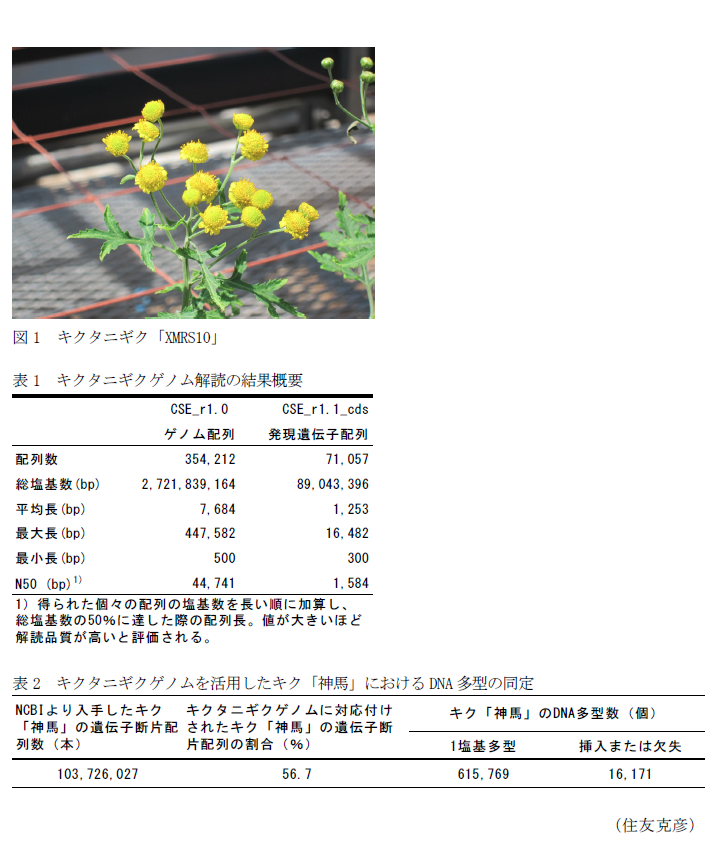
- 広島大学において自家和合性の野生系統から自殖によって作出された純系キクタニギク系統「XMRS10」(図1)を材料とし、超並列シークエンシング技術と大型計算機を用いて解読した27.2億塩基のゲノム配列CSE_r1.0は、キクタニギク全ゲノムの89%に相当する。解読結果の質を示す指標であるN50は44,741塩基であり、全ゲノムの概要情報として十分な精度をもつ(表1)。
- キクタニギク系統「XMRS10」から発現遺伝子を抽出し解読した配列を、前項のゲノム配列と合わせてさらに再構築し、300塩基以上の配列のみを取りまとめたデータセットCSE_r1.1._cdsにおいて、推定される遺伝子数は71,057である(表1)。
- キクタニギク全ゲノムとキク品種のゲノム断片配列を比較し、キクゲノム配列上のDNA多型を検出できる。例えば、NCBI(National Center for Biotechnology Information)より入手したキク「神馬」の遺伝子断片情報を用い、キクタニギクゲノムへ対応付けして再構成されたキク「神馬」の配列情報より615,769個の1塩基多型および16,171個の挿入または欠失が同定できる(表2)。これらDNA多型はDNAマーカーとして活用できる。
成果の活用面・留意点
- 配列情報や配列内の変異を染色体上に並べた連鎖地図は、かずさDNA研究所のサーバーからデータベースとして公開している。(http://mum-garden.kazusa.or.jp/)
- ゲノム情報を用いたキクの育種が進むと期待される。
具体的データ
その他
- 予算区分:交付金、委託プロ(次世代ゲノム(DHR5))、競争的資金(科研費)
- 研究期間:2015~2018年度
- 研究担当者:
住友克彦、平川英樹(かずさDNA研)、久松完、永野聡一郎(森林総研)、白澤健太(かずさDNA研)、樋口洋平(東京大)、草場信(広島大)、腰岡政二(日本大)、中野善公、八木雅史、山口博康、谷口研至(広島大)、中野道治(広島大)、磯部祥子(かずさDNA研) - 発表論文等:Hirakawa et al. (2019) DNA Research 26:195-203
