農業生物のマルチオミクスデータ解析支援アプリケーション「CoreNet+」
要約
「CoreNet+」とは、研究者が集積した農業生物の多様なオミクス情報(品種特性情報、遺伝子発現情報、代謝物蓄積情報等)を解析して並列的にデータベース化し、ウェブインターフェースからデータマイニング可能なソフトウェアパッケージである。
- キーワード : バイオインフォマティクス、オミクスデータ、ゲノム、遺伝子、ビッグデータマイニング
- 担当 : 基盤技術研究本部・高度分析研究センター・ゲノム情報大規模解析ユニット
- 代表連絡先 :
- 分類 : 研究成果情報
背景・ねらい
近年、農業生物の個体レベルの網羅的分子情報(オミクスデータという)をハイスループットに一斉分析し、取得する技術が進展している。オミクスデータにはゲノム全体の遺伝子発現データであるトランスリプトーム情報や様々な代謝物の蓄積情報を網羅したメタボローム情報の他、タンパク質の蓄積・修飾情報であるプロテオーム、品種や遺伝資源の形質情報であるフェノーム、微生物叢情報であるマイクロバイオーム、DNA多型情報であるバリオーム等が存在する。これらは温度や湿度などの生育環境情報と紐づけられる場合もあり、統合マルチオミクスデータとして集積されるケースが多くなってきている。その大規模に集積されたオミクスデータ(ビッグデータ)により、因子間の関連性が明らかになり、遺伝子や代謝物の新しい機能が発見されたり、品種改良や生産技術の向上に役立つ情報をマイニングするためのデータ解析基盤に用いられたりするなど、その重要性が増している。CoreNet+は、それらに貢献するために多様なオミクスデータの情報解析とデータベース化をワンコマンドでバッチ処理的に実行することができ、且つ、ウェブブラウザやAPIからデータ検索とマイニングが可能なアプリケーションである。
成果の内容・特徴
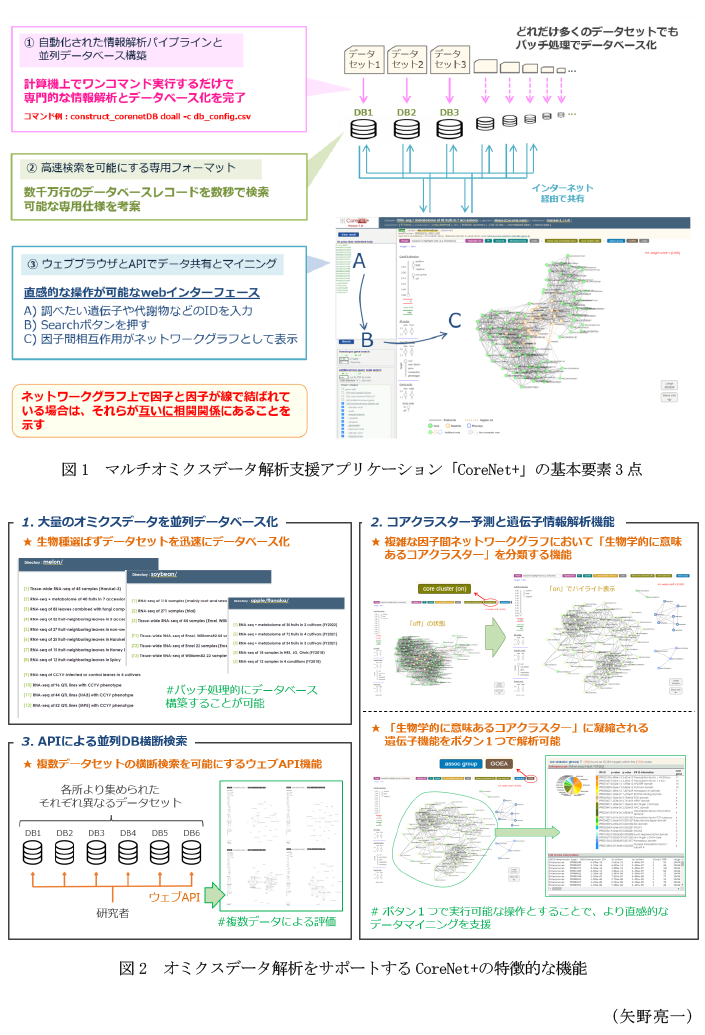
- 本アプリケーションは、①オミクスデータ解析とデータベース構築の情報処理パイプライン(プログラムの集合体)、②高速検索を可能にする独自のデータベースフォーマット、③データアクセス・マイニング用のウェブインターフェース、の3点を1つのパッケージとしてまとめたものである(図1)。
- ①の情報処理は数値行列データ(オミクスデータ)とゲノムリファレンス情報(Fasta、GFF形式)を入力とする。対象生物のゲノム全体の遺伝子情報を統合する形で因子間相互作用(例えば代謝物と遺伝子の関連等)を算出し、さらに高速検索に適したフォーマットでデータベース②を生成する。フレキシブルなデータ管理を可能にするために、「1データセット=1データベース方式」を採用している(図1右上)。
- ①は生物種を問わず、計算機上で、ワンコマンドで実行可能としており、データセットが多岐に渡っている場合でもバッチ処理的にデータベース化することが可能である(図2左上)。
- ウェブインターフェース③を介して遺伝子IDや代謝物ID等のクエリー情報を送信することで、構築されたデータベース②に対して検索を実行する。因子間の関連をネットワークグラフとして取得し、情報分析することができるが、インターフェースの操作パネルにてスレッシュホールドを変更したり、より重要な相互作用グループ(コアクラスター)をハイライトしたりすること等が可能である(図1右下、図2右)。
- ウェブAPI機能によって、並列的に構築されたデータベースを横断的に検索することも可能である(図2左下)。
成果の活用面・留意点
- 本アプリケーションは数値行列化されたオミクスデータを念頭に置いている。すなわち、トランスリプトーム(発現遺伝子情報)、メタボローム(代謝物蓄積情報)、プロテオーム(タンパク蓄積情報)、フェノーム(品種特性情報など)、マイクロバイオーム(細菌叢情報)、DNA多型情報及びそれらの混合データを対象としている。
- 主としてオミクスデータにトランスリプトームが含まれる場合を想定しており、データベース構築コマンドラインにてゲノムリファレンス情報を指定することで、ウェブインターフェースにおけるGOタームエンリッチメント解析機能等が使用可能となる。
- 本アプリケーションはインターネット環境とウェブサーバーの使用を前提しているが、ローカルネットワークあるいは仮想環境でのスタンドアロンでの利用も可能である。
- 本アプリケーションの使用ケースとしては、特定の二次代謝成分に関連する遺伝子の特定や、多重コピーした重複遺伝子の機能分類、特定の品種が持つ遺伝子相互作用ネットワークの比較解析等が挙げられる。
具体的データ
その他
- 予算区分 : 文部科学省(戦略的創造研究推進事業)、農林水産省(ムーンショット型農林水産研究開発事業)
- 研究期間 : 2018~2022年度
- 研究担当者 : 矢野亮一
- 発表論文等 : 矢野、特願(2022年9月15日)
