多品種・系統群の集団構造や近縁性を解析し結果をウェブ・アプリケーション化するパイプライン「PSReliP」
要約
「PSReliP」とは、ゲノムワイド関連解析(GWAS)やゲノミックセレクション等の多品種・系統群のゲノム解析において重要な、集団構造や近縁性の解析および解析結果を可視化するアプリケーションの構築までを一括で実行可能なパイプラインである。
- キーワード : バイオインフォマティクス、集団ゲノミクス、ウェブ・アプリケーション
- 担当 : 基盤技術研究本部・高度分析研究センター・ゲノム情報大規模解析ユニット
- 代表連絡先 :
- 分類 : 研究成果情報
背景・ねらい
多品種・系統のゲノム情報を解析し、特定の表現形質と遺伝子との関連を同定する手法であるゲノムワイド関連解析(GWAS)や、表現形質を予測するゲノミックセレクション(GS)において、品種・系統群の集団構造や近縁性は、解析あるいは予測結果に大きな影響を及ぼす因子であることから、事前に十分検討しておくことが重要である。それらを解析あるいは解析結果を可視化するツールは多く存在するが、解析から可視化までを一括で行えるようなツールは存在しておらず、その開発が望まれている。「PSReliP」(ピーエスレリピイ)は、一般的なゲノム解析ツールであるPLINKと、インタラクティブなウェブ・アプリケーションを構築するためのフレームワークであるShiny®を利用し、集団構造や近縁性の解析から、解析結果をインタラクティブに閲覧できるウェブ・アプリケーションの作成までを自動実行するパイプラインである。PSReliPの実行にはバイオインフォマティクスの専門的な知識は不要であり、Linux®環境さえあれば誰でも簡単に利用することができる。
成果の内容・特徴
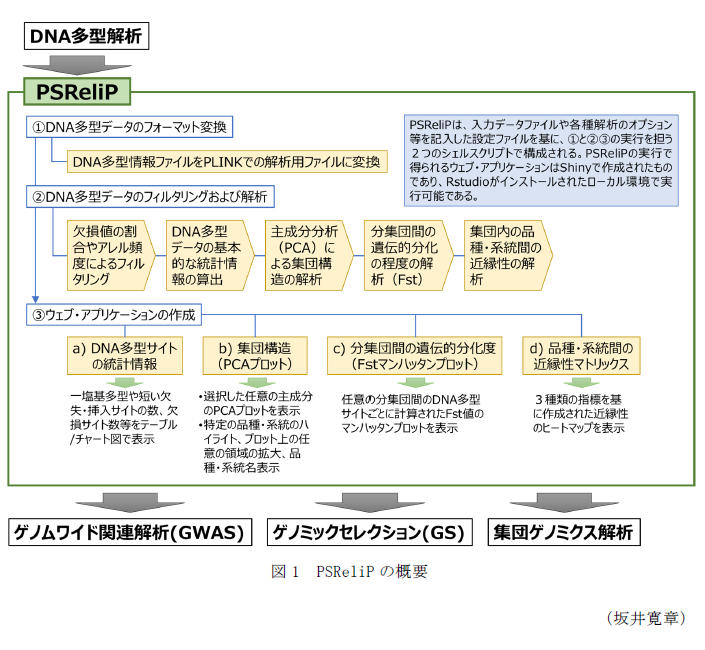
- PSReliPは、①DNA多型データのフォーマット変換、②DNA多型データのフィルタリングおよび解析、③ウェブ・アプリケーションの作成、の3段階で構成され、①の実行と②および③の実行を担う2つのシェルスクリプトとして実装されている(図1)。利用者は、Linux®サーバー上で入力データファイルや各種解析のオプション等を記入した設定ファイルを作成し、2つのシェルスクリプトを順次実行することで結果を取得できる。ウェブ・アプリケーションはShiny®で作成されたものであり、ウェブ・サーバーが無くても、RStudio®がインストールされたローカル環境で実行可能である。
- ウェブ・アプリケーション上では、4種類のデータを画面上でタブを切り替えながら閲覧できるようになっている。a)DNA多型サイトの統計情報タブでは、各品種・系統の一塩基多型(SNP)や短い欠失・挿入サイトの数や、欠損サイト数等をテーブルかチャート図で閲覧できる。b)集団構造タブでは、任意の主成分を選択して主成分分析(PCA)のプロットを表示できるほか、特定の品種・系統をハイライトしたり、プロット上の任意の領域を拡大し品種・系統名を表示したりすること等ができる。c)分集団間の遺伝的分化度タブでは、任意の分集団間のDNA多型サイトごとに計算されたFst値のマンハッタンプロットを閲覧できる。Fst値とは、分集団間の遺伝的な分化度を測る指標であり、0~1の値を取り、1に近いほど分集団が分化していることを意味する。d)品種・系統間の近縁性マトリックスタブでは、3種類の指標を基に作成された近縁性のヒートマップを閲覧できる。
- 統計情報や主成分分析結果等のデータはアプリケーション画面上からダウンロード可能であり、図については一部のインタラクティブな機能を維持したままHTMLファイルとして保存することができる。
成果の活用面・留意点
- PSReliPは、GWASやGS等の高度なゲノム解析の支援を目的として開発されたが、主成分分析や近縁性の解析は一般的であるほか、PSReliPではDNA多型サイトの基本的な統計情報も閲覧できることから、多品種・系統のゲノム情報を対象にした集団ゲノミクス解析に広く活用できるツールである(図1)。
- PSReliPの実行にはLinux®環境とゲノム解析ツールであるPLINKがインストールされている必要があるが、PLINKによる解析自体は自動実行されるため、バイオインフォマティクスの専門知識を持たない研究者であっても利用できる。
- PSReliPはGitHub®上で公開しており、誰でもダウンロードできる。
具体的データ
その他
- 予算区分 : 農林水産省(みどりの食料システム戦略実現技術開発・実証事業のうち農林水産研究の推進、戦略的プロジェクト研究推進事業:民間事業者等の種苗開発を支える「スマート育種システム」の開発)
- 研究期間 : 2018~2022年度
- 研究担当者 : 坂井寛章、ソロビヨワ・イエレナ
- 発表論文等 :
- Solovieva E. and Sakai H. (2023) BMC Bioinformatics 24:135
- https://github.com/solelena/PSReliP
