時系列発現量データを利用した遺伝子調節ネットワークの推定
要約
時系列遺伝子発現量の差分に基づく、多変量解析手法と総当たりの単回帰分析を用いて遺伝子制御関係を推定する手法である。
- キーワード : 遺伝子解析、遺伝子制御、ネットワーク、時系列データ
- 担当 : 基盤技術研究本部・農業情報研究センター・AI研究推進室・確率モデルユニット
- 代表連絡先 :
- 分類 : 研究成果情報
背景・ねらい
あらゆる細胞プロセスは、遺伝子間の相互作用で構成され、ネットワークとしてモデル化でき、遺伝子調節ネットワークなどと呼ばれている。遺伝子調節ネットワークを推定することは、生命現象の基本メカニズム解明に必要な基盤技術となるだけでなく、生物由来の有用物質の生産に関わる遺伝子(有用遺伝子)の発見やその効率的な利用にも役立つ。このような有用遺伝子の探索には、異なる条件下で発現量が異なる遺伝子を統計的に検出する方法や互いに似た発現変動をする遺伝子同士をクラスタリングによって検出する方法などが利用されている。このなかで、遺伝子調節ネットワークを利用することで、有用遺伝子の制御メカニズムを推定でき、その発現調節遺伝子を正確に同定することができるようになる。近年、遺伝子発現(発現量)計測技術の進歩により、遺伝子の時系列発現変動データが安価かつ大量に得られるようになった。遺伝子調節ネットワークの推定において、このような大量のデータが必要不可欠であるが、対象となる遺伝子の数が増えると、遺伝子間の組み合わせの数が指数関数的に増えてしまうため、既存手法では有限時間内で遺伝子調節ネットワークを推定することが極めて困難となり得る。そのため、効率的な探索アルゴリズムの開発が求められている。
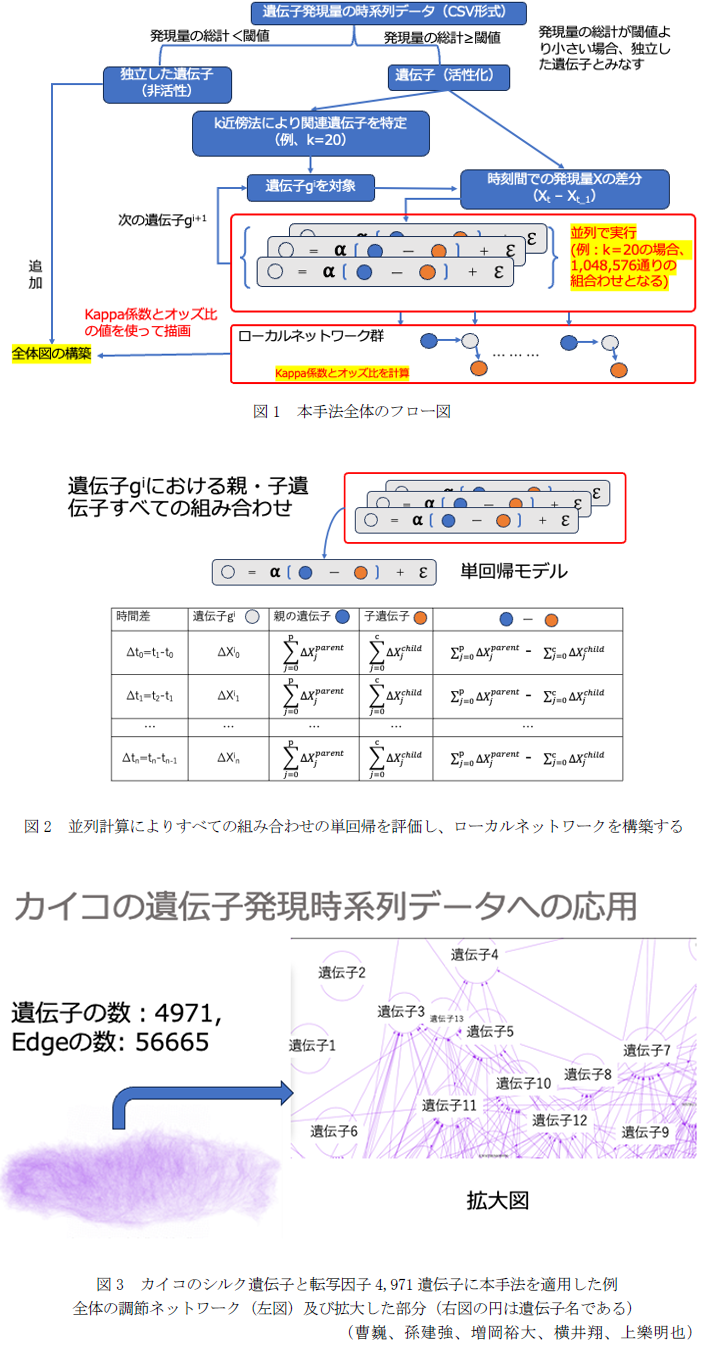
そこで本研究では、時系列遺伝子発現量に基づく、多変量解析手法(k近傍法)により関連遺伝子群を特定し、この遺伝子間のネットワーク構造(本研究でローカルネットワークと呼ぶ)を総当たりの回帰分析を並列計算により実行して調節関係強度を評価する。そして、調節関係強度を用いて、調節ネットワークの全体図(有向非巡回グラフ)を推定する手法を提案する。この全体図は、本手法の出力として、既存のネットワーク可視化プラットフォームにも適用できるように、データ記述言語であるdot言語を使って描画する。また、本手法をカイコの絹糸腺から得られた遺伝子発現量の時系列データに適用することで、シルク合成に関する遺伝子間の調節ネットワークを明らかにすることが可能である。
成果の内容・特徴
- 本研究で提案する手法は、(1)発現量の総計による活性化・非活性化の振り分け、(2)k近傍法による関連遺伝子群の特定、(3)発現量の時点間の差分に対する全ての組み合わせの単回帰の実行によるローカルネットワーク群の構築、(4)Kappa係数とオッズ比の値による影響度評価と全体のネットワークの構築によって構成される(図1)。
- ローカルネットワークの構築において、情報理論の観点に基づき、ある遺伝子の発現量の変化を、その遺伝子の上流および下流にある遺伝子群の総変化の結果と考えて単回帰モデルで評価し、最適なローカルネットワークを構築する。なお、この処理では大量の組み合わせを処理する必要があるが、大型計算機などを活用した並列計算による高速化が可能である(図2)。
- カイコのシルク生産遺伝子と転写因子4,971遺伝子の時系列発現量データに本手法を適用し、調節ネットワークを構築した例を図3に示す。遺伝子発現量は、TPM(Transcripts Per kilobase Million)によって正規化している。なお本手法では、遺伝子発現量が統計学上の特定の確率分布に従う仮定を置く必要はない。
成果の活用面・留意点
- 本手法はカイコの絹糸腺に限らず、様々な生物種の遺伝子発現系に応用可能で、マイクロアレイ時系列データ、単細胞RNA-seqを含む様々なRNA-seqの時系列データに適応できる。
- 細胞内で起きている生命現象のメカニズムを理解するためには、推定した遺伝子間の大規模な調節ネットワークが不可欠であり、このような知見は、生物の物質生産性に寄与する遺伝子群の推定や、人為的制御による複雑・高機能な物質の生産にも貢献できる。
- 本手法では、ローカル遺伝子調節ネットワークの構築のために近隣遺伝子の数を事前に設定する必要がある。大きな数値を設定することで、より正確なローカルネットワークを探索できるが、計算量が膨大になることから計算機リソースの制約により実行できない可能性がある。
具体的データ
その他
- 予算区分 : 交付金、文部科学省(共創の場形成支援プログラム)
- 研究期間 : 2022~2023年度
- 研究担当者 : 曹巍、孫建強、増岡裕大、横井翔、上樂明也
- 発表論文等 : 曹ら、特願(2024年5月8日)
