いも類等同質倍数体作物における全ゲノム配列を用いた量的形質遺伝子座迅速検出法
要約
バレイショ、サツマイモ等の同質倍数体作物の交雑集団において、目的形質の有無で分別した個体群をそれぞれまとめて全ゲノム配列を取得し、各相同染色体に固有の一塩基多型を抽出し、その出現頻度を比較することで、連鎖地図を作成せずに、迅速に量的形質遺伝子座(QTL)を検出できる。
- キーワード:同質倍数体、バレイショ、サツマイモ、量的形質遺伝子座(QTL)、全ゲノムシーケンス
- 担当:作物研究部門・作物デザイン研究領域・作物デザイン開発グループ
- 代表連絡先:
- 分類:研究成果情報
背景・ねらい
バレイショとサツマイモはそれぞれ同質四倍体、同質六倍体のゲノムをもち、複雑な遺伝様式を示すため、優良個体の選抜を効率化するDNAマーカーの開発や利用が限定的で、新品種開発に多大な時間と労力を必要とする。
そこで、本研究では、近年低価格で大量の塩基配列データを取得可能となった次世代シーケンス技術と高性能コンピューターを活用して、同質倍数体作物において全ゲノム配列データから迅速にQTLを検出する方法を開発し、DNAマーカー作製の加速化に資する。
成果の内容・特徴
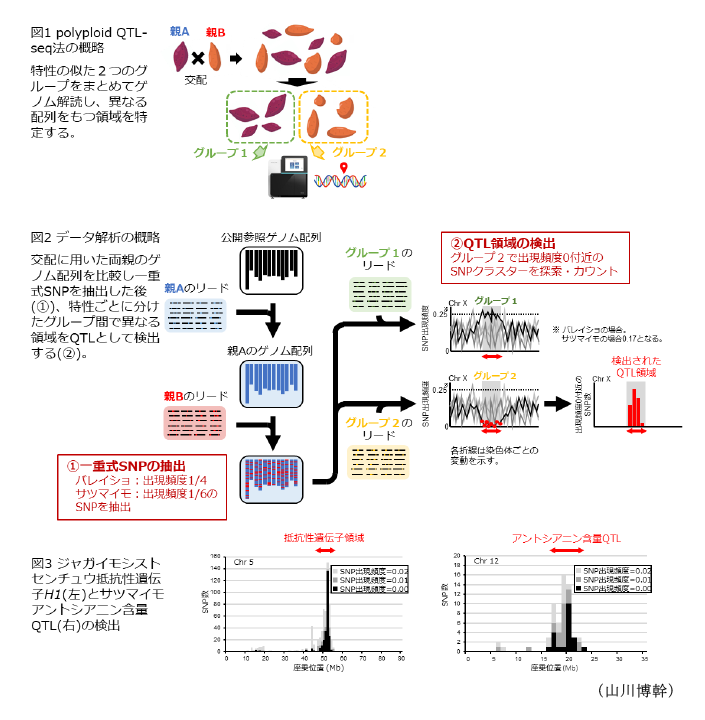
- 本法の概略を図1に示す。特性の異なる2品種(親Aおよび親B)の交配により後代集団を作成し、特性の似た2つの個体群をまとめて(グループ1およびグループ2)、それぞれゲノム配列を解読し、両個体群において異なる塩基配列をもつ染色体領域をQTLとして特定する。
- データ解析は図2に示す手順で行う。【①一重式一塩基置換(SNP)の抽出】一重式SNPとは、1本の相同染色体のみに存在し、交雑後代で単純な分離を示すSNPのことであり、これのみを抽出してQTL解析に利用する。交配両親についてそれぞれ100 Gbp程度の全ゲノム配列データを取得し、公開参照ゲノム配列を一方の親(親A)のSNPで置き換えたゲノム配列を作成する。つぎに、同配列ともう一方の親(親B)の全ゲノム配列データとを照合し、四倍体のバレイショでは出現頻度が1/4、六倍体のサツマイモでは1/6のSNPを一重式SNPとして抽出する。【②QTL領域の検出】:交雑一代(F1)集団において、目的とする形質を示す個体群(グループ1)と示さない個体群(グループ2)に分けて、それぞれ個体をまとめて全ゲノム配列を取得し(図1)、①で抽出した一重式SNPの出現頻度を比較すると、目的形質を制御するQTLの近傍のSNPのみが、両群間で異なる出現頻度を示す。このうち、グループ2で、SNP出現頻度が0付近となるSNPがクラスター状に存在する領域にQTLがあると判断できる。
- 本法を、ジャガイモシストセンチュウ抵抗性H1遺伝子、およびサツマイモ塊根の紫色素アントシアニン蓄積に適用すると、それぞれ5番染色体および12番染色体に単一のQTL領域が検出される(図3)。QTL近傍のSNPを用いてPCRマーカーを設計すると、供試した解析集団で完全連鎖を示すDNAマーカーを作製できる。
- 本法では、シーケンス解析のための煩雑なDNA調製や、高度な遺伝連鎖地図作成技術を必要とせず、F1集団の形質評価から最短で3か月程度でQTLを検出できる。また、目的形質を別の品種に導入する場合にも、QTL領域に見いだされる多数のSNPを利用して、交配組み合わせに応じて新たに連鎖DNAマーカーを作製することが可能である。
成果の活用面・留意点
- 本法は同質倍数体向けpolyploid QTL-seq法として公表され、公益財団法人岩手生物工学研究センターで公開されるQTL-seqパイプライン(https://genome-e.ibrc.or.jp/resource/mutmap)とマイクロソフトExcelを用いることで、実施可能である。
- 本法は、Linux環境に対応できるコンピューターを用いる。バレイショおよびサツマイモでの解析に必要な仕様として、Macintoshを用いる場合、メモリ64MB、HDD 2TBを備える必要がある。
- 参照ゲノム配列が公開されている同質倍数体作物(バラ、ブルーベリー、サトウキビ等)についても、倍数性に応じて一重式SNPを抽出するための閾値条件を変更することで本法が適用可能である。
具体的データ
その他
- 予算区分:交付金
- 研究期間:2020~2021年度
- 研究担当者:山川博幹、ハク エムダドウル、田中勝、浅野賢治、下坂悦生、赤井浩太郎、岡本智史、片山健二、田宮誠司、高木宏樹(石川県立大)
- 発表論文等:Yamakawa H. et al. (2021) Plant Biotechnol. J. 19:2040-2051
