ゲノム全体をカバーしたタマネギDNAマーカーセットを効率的に作成する手法
要約
品種間のDNA多型がある発現遺伝子の中からゲノム全体に均一に分布するように遺伝子を選び、それらの配列情報を用いてDNAマーカーセットを作成する。この手法で作成したDNAマーカーセットは8本の染色体に均等に分布し、タマネギのゲノム全体の9割以上をカバーする。
- キーワード : タマネギ、発現遺伝子、DNA多型、DNAマーカー
- 担当 : 野菜花き研究部門・野菜花き育種基盤研究領域・育種技術開発グループ
- 代表連絡先 :
- 分類 : 研究成果情報
背景・ねらい
ゲノム全体をカバーしたDNAマーカーセットは、多検体の解析集団での遺伝子型取得およびそれらを用いた目的形質の遺伝解析に有効である。多くの作物において、DNAマーカーセットの作成には全ゲノム解析から取得したDNA多型情報を利用している。しかし、タマネギのゲノムサイズは野菜の中でも最大級(16 Gb、イネの約40倍、トマトの16倍)であり、全ゲノム解析での多型検出にはコストを要する。そこで、全ゲノム解析の約1/10のコストで多型情報を取得できる発現遺伝子解析(RNA-seq)を利用し、ゲノム全体をカバーしたタマネギDNAマーカーセットを効率的に作成する方法を開発する。
成果の内容・特徴
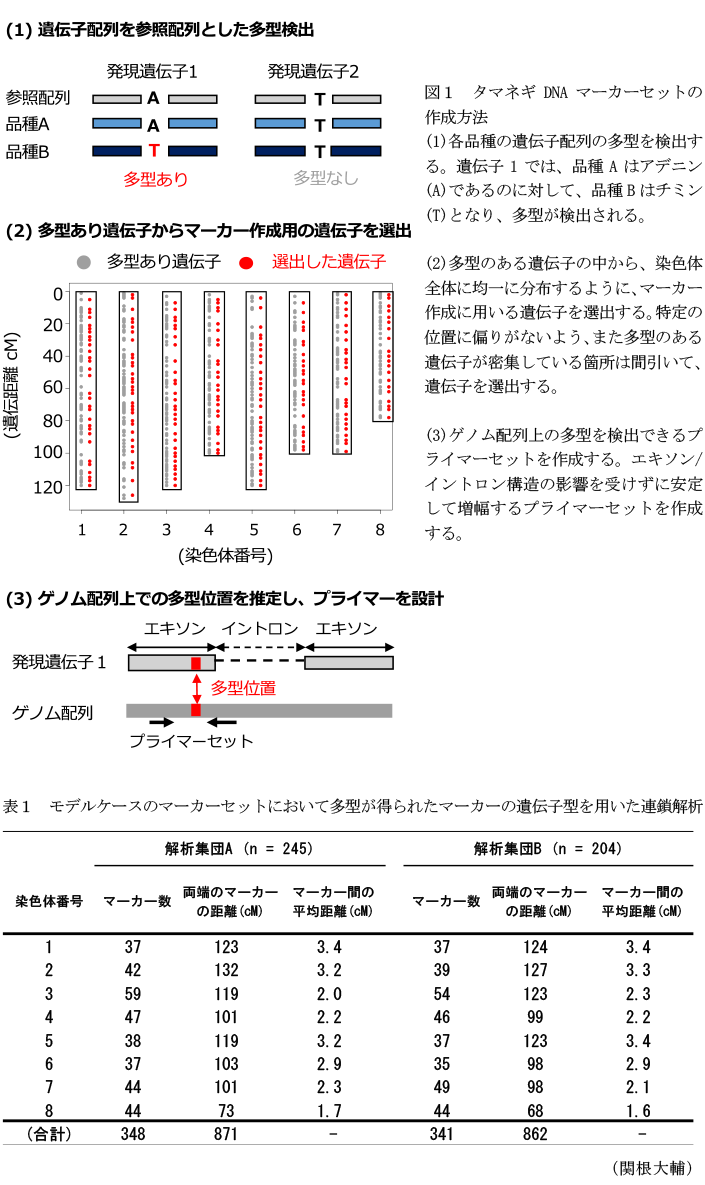
- 本タマネギDNAマーカーセットの作成方法は、3つの手順によって構成される(図1)。 (1)各品種のRNA-seqの解析結果と既存の遺伝子配列(参照配列)との比較により、品種間の多型の検出および多型のある発現遺伝子を抽出する。 (2)先行研究(Fujito et. al., 2021, BMC Genomics., 22, 481)で推定された染色体上の遺伝子の位置情報を用いて、多型のある遺伝子の中から、染色体全体に均一に分布するようにマーカー作成用の遺伝子を選出する。 (3)ゲノムDNAを鋳型としたPCRでエキソン/イントロン構造の影響を受けないよう、選出した遺伝子に対応するゲノム配列を抽出し、多型位置を増幅するプライマーセットを設計する。
- モデルケースとして作成した441個のDNAマーカーセットでは、2つの集団について約8割のマーカー(348, 341マーカー)で多型が得られる。多型が得られたマーカーはゲノム全体に均一に分布する。第1-8染色体ごとのマーカー数は37-59個、各染色体のマーカー間の平均距離は1.6-3.4 cMである(表1)。
- 2集団におけるDNAマーカーセットの遺伝距離の累計は871 cMと862 cMであり、先行研究で報告された1449マーカーでの遺伝距離の累計(937 cM)の92 %以上をカバーする(表1)。
成果の活用面・留意点
- タマネギDNAマーカーセットは、多検体でのゲノム全体の遺伝子型の効率的な取得を可能とし、目的形質の遺伝解析や目的形質と密接に関連した選抜マーカーの開発に有用である。
- モデルケースとしたDNAマーカーセットは、2品種間の多型情報を用いて作成しており、この品種間の交配後代集団の遺伝子型解析に有用である。このDNAマーカーセットを別の品種間の交配後代集団の遺伝子型解析に用いる場合、多型が得られるマーカー数が減少することが予想される。解析対象に合わせて、新たにDNAマーカーセットを作成する必要がある。品種間の多型解析の結果から、モデルケースのDNAマーカーセットの中で多型が得られるマーカーがあれば、それらを選出する。モデルケースのDNAマーカーセットで多型が得られない領域がある場合は、本手法により新たにDNAマーカーを設計し、追加する。
- 本マーカー作成方法の実行には、バイオインフォマティクスの知識を要する。マーカー作成に用いた複数のコンピュータープログラム、参照配列として用いた遺伝子配列、作成したDNAマーカーセットのプライマー配列の詳細は下記の発表論文に記載する。
- RNA-seq には、各品種の葉から抽出したRNAを用いる。
具体的データ
その他
- 予算区分 : 文部科学省(科研費)、その他外部資金(SIPII)、その他外部資金(戦略的国際共同研究推進委託事業)
- 研究期間 : 2018~2022年度
- 研究担当者 : 関根大輔、奥聡史、布目司、平川英樹(かずさDNA研)、辻村真衣(龍谷大)、寺地徹(京都産業大)、豊田敦(遺伝研)、執行正義(山口大)、佐藤修正(東北大)、塚﨑光
- 発表論文等 : Sekine D. et al. (2022) DNA research doi:10.1093/dnares/dsac020
