ネギハモグリバエのバイオタイプ識別が可能な核DNAマーカー
要約
PCR-RFLP(制限酵素切断断片長多型)法を用いた核DNAマーカーにより、ネギハモグリバエの2種類のバイオタイプを識別できる。本識別法により、既存のミトコンドリアDNAマーカーでは識別できなかったバイオタイプ間交雑個体も識別可能となり、全国の病害虫防除所等で活用できる。
- キーワード : ネギハモグリバエ、核DNAマーカー、バイオタイプ、PCR-RFLP法
- 担当 : 野菜花き研究部門・野菜花き育種基盤研究領域・素材開発グループ
- 代表連絡先 :
- 分類 : 研究成果情報
背景・ねらい
ネギハモグリバエはネギ類の重要害虫である。近年、従来のバイオタイプAより甚大な被害をもたらすバイオタイプBの発生が全国的に拡大している。バイオタイプBと疑われる被害が新たに発生した場合、特殊報発令のために確実な同定が必要となるが、バイオタイプ識別に用いられているミトコンドリアDNAマーカーでは母親由来の遺伝子型しか確認できず、同定には不十分である。本研究ではバイオタイプ識別が可能な核DNAマーカーを開発し、既存のミトコンドリアDNAマーカーとの併用による確実なバイオタイプ識別を可能にすることで、全国の病害虫防除所等でのバイオタイプ同定に貢献する。
成果の内容・特徴
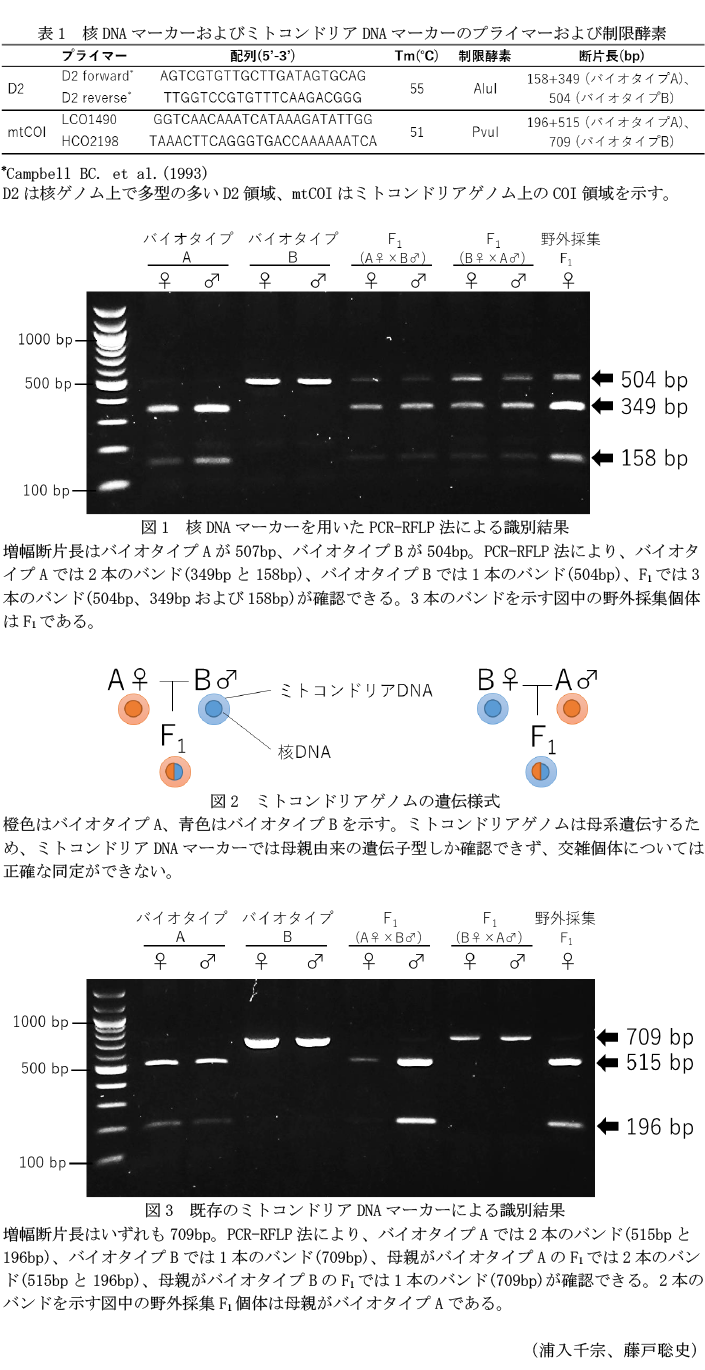
- 核ゲノム上で多型が多いことから昆虫の系統分化推定に使用されるD2領域に設計されたプライマー(Campbell BC. et al.,1993、表1)を用いてシーケンスを行うと、バイオタイプAに由来する増幅断片は制限酵素AluI認識部位を有することを確認できる。DNA断片をAluIで処理し、アガロースゲル電気泳動で分画すると、2つのバイオタイプおよびその交雑個体F1に由来するDNA断片を識別できる(図1)。
- ミトコンドリアゲノムは母系遺伝するため(図2)、既報のミトコンドリアDNA上のCOI領域に設計されたプライマー(表1)で増幅したDNA断片をPvuIで処理し、アガロースゲル電気泳動で分画すると、2つのバイオタイプに由来するDNA断片を識別でき(Tokumaru S. et al.,2021)、母親由来の遺伝子型を確認できる(図3)。核DNAマーカーと既報のミトコンドリアDNAマーカーの併用により、交雑個体の母親由来の遺伝子型を確認できる(図1、3)。
成果の活用面・留意点
- 本識別法はネギハモグリバエが発生する全国の都道府県病害虫防除所において活用できる。
- ネギハモグリバエのバイオタイプ識別のための核DNAマーカーであるため、他のハモグリバエ類を同定する目的では使用できない。
- 既存のミトコンドリアDNAマーカーと組み合わせることで正確なバイオタイプ識別が可能となり、ネギハモグリバエのバイオタイプ間交雑に関する研究の加速化につながる。
具体的データ
その他
- 予算区分 : 交付金
- 研究期間 : 2020~2021年度
- 研究担当者 : 浦入千宗、藤戸聡史、上杉龍士
- 発表論文等 : Urairi C. et al. (2022) Appl. Entomol. Zool. 57:313-322
