ウンシュウミカンのゲノムライブラリの構築
※アーカイブの成果情報は、発表されてから年数が経っており、情報が古くなっております。
同一分野の研究については、なるべく新しい情報を検索ください。
要約
ウンシュウミカン「宮川早生」のBACライブラリを構築した。その挿入断片の平均長は約133kbで、全クローンの総和は推定ゲノムサイズの13.3倍以上である。 新規DNAマーカーの開発、プロモータの解析、物理地図の作製などに利用できる。
- キーワード:カンキツ、ゲノム、ライブラリ、BAC、クローニング
- 担当:果樹研・カンキツ研究部・遺伝解析研究室(カンキツゲノム研究チーム)
- 連絡先:成果情報のお問い合わせ
- 区分:果樹・育種
- 分類:科学・参考
背景・ねらい
カンキツ育種では品種育成に長期間を要するため、事前に果実特性などを判定することのできる早期判別技術の開発が求められている。DNAマーカーを利用した遺伝連鎖地図の作製と早期選抜は有望な手法であるが、選抜したい形質と連鎖するDNAマーカーの開発に長期間を要すること、果樹では連鎖地図でのDNAマーカーを高密度化しにくいという問題がある。 重要な形質と強く連鎖するDNAマーカーの開発や、すでに開発されたDNAマーカーを他の交雑集団に確実に移植するためには、カンキツのゲノム情報を直接利用する手法が有効と考えられる。そこで、バクテリア人工染色体(BAC)ベクタを利用して、ウンシュウミカンのゲノムライブラリを作製した。
成果の内容・特徴
- ウンシュウミカン「宮川早生」の全DNAを断片化したのちBACベクタ(pECBAC1)に組み込んだBACライブラリを作製した。作製したライブラリは全36,864クローンからなり、マルチウェルプレートに個別に分注されている。各プレートのクローンを一つにまとめたプールプレートと、さらにこれを一つのプレートにまとめたスーパープールプレートを作製することで、PCR法によるスクリーニングを簡略化することが可能である。
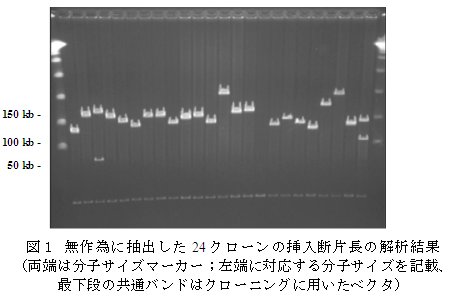
- 無作為に取り出した24個のクローンについて、パルスフィールドゲル電気泳動法で検定した挿入断片の平均鎖長は132.7kbであり、この結果から本ライブラリの全長はウンシュウゲノムの推定値の13.3倍に相当すると推定される(図1)。
- 単一遺伝子由来と考えられる既存のDNAマーカー15個と対応するBACクローンをPCR法でスクリーニングする手法では、それぞれ3~37個の対応するクローンが得られている。この結果から推定される本ライブラリ中のDNA断片の総和は、ウンシュウゲノムの推定値の17.3倍相当量と考えられる(表1)。
成果の活用面・留意点
- 2通りの手法で評価した本BACライブラリのウンシュウゲノム相当量は13.3から17.3倍と推定され、ウンシュウミカンの全ゲノム領域にわたる物理地図作製にも利用可能である。
- 既存のDNAマーカーと対応するクローンを選抜してその塩基配列を解析することで、ウンシュウミカンの連鎖地図と対応する領域の詳細な情報を知ることができる。その結果、ゲノムの塩基配列情報を直接利用したDNAマーカーの開発が容易となる。
- cDNAと対応するゲノムDNA断片を単離して解析することで、遺伝子プロモータなどの情報が得られ、遺伝子の発現制御機構の解析に貢献できる。
- 葉緑体DNAの5箇所のDNAマーカーを用いた検定から、本BACライブラリ中の葉緑体DNAの混入率は約1.8%と推定される。
具体的データ

その他
- 研究課題名:カンキツのDNAマーカーの作出と高密度連鎖地図の作製
- 課題ID:09-02-07-*-07-04
- 予算区分:DNAマーカー
- 研究期間:2003~2004年度
- 研究担当者:島田武彦、遠藤朋子、藤井浩、西川芙美恵、大村三男(静岡大)、清水徳朗
