カンキツ発現遺伝子の機能推定のための自動処理システム
※アーカイブの成果情報は、発表されてから年数が経っており、情報が古くなっております。
同一分野の研究については、なるべく新しい情報を検索ください。
要約
コンピュータ・システム(パイプライン)によりカンキツで発現する大量の遺伝子の塩基配列を配列の類似性に基づいて分類し、既知の遺伝子配列との類似性から遺伝子機能を推定する作業(アノテーション)を自動的かつ連続的に実行することができる。
- キーワード:カンキツ、ゲノム、塩基配列、発現遺伝子、アノテーション、パイプライン
- 担当:果樹研・カンキツ研究部・遺伝解析研究室(カンキツゲノム研究チーム)
- 連絡先:成果情報のお問い合わせ
- 区分:果樹・育種
- 分類:科学・参考
背景・ねらい
発現遺伝子の解析は、遺伝子の単離やDNAマーカーの作成、マイクロアレイの設計など広範に利用できるため、重要なゲノム解析手法である。また、近年、塩基配列解読のコストが下がり、発現遺伝子の解読が進み、公共データベースにも多数のEST(発現遺伝子の断片的な塩基配列)情報が公開されている。そこで、大量の発現遺伝子の塩基配列を自動的かつ連続的に処理し、遺伝子機能を迅速に推定するためのアノテーション・パイプラインが不可欠である。
成果の内容・特徴
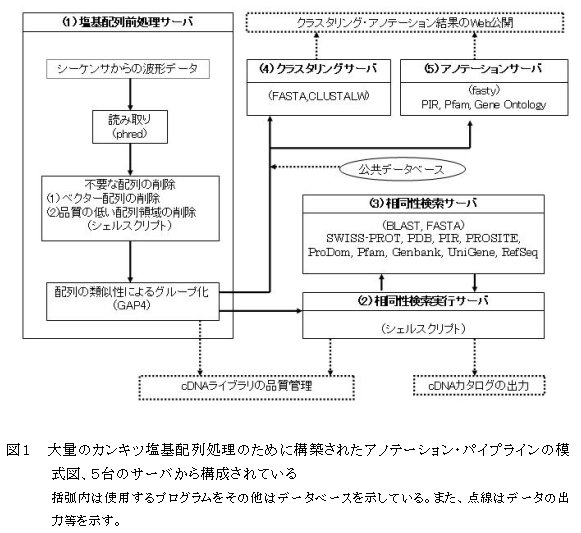
- 本システムは、シーケンサから出力された波形データから塩基配列情報を読み取り、クラスタリング及びアノテーションまでを自動的に行う。(図1)。
- サーバは、以下の5つである。
(1)塩基配列前処理サーバ:シーケンサから出力される波形データを読み取り、不要配列の除去、配列の類似性の検
定、ローカルの相同性検索サーバに対する相同性検索といった波形データ処理の一連の作業を連続的・自動的に
行う。
2)相同性検索実行サーバ:相同性検索サーバを制御して相同性検索を自動的かつ連続的に行う。
3)相同性検索サーバ:主要な9つの遺伝子データベース(SWISS-PROT等)をサーバにダウンロードし、これらに対して
BLASTとFASTAを用いて検索を実行する。
4)クラスタリングサーバ:10万のオーダーの塩基配列を類似性に基づいて分類する。
5)アノテーションサーバ:発現遺伝子の塩基配列に対して、自動的に3つの遺伝子データベース(PIR、Pfam、Gene
Ontology)によるアノテーションを行う。 - クラスタリングサーバ及びアノテーションサーバの計算結果をWeb経由でブラウザによって閲覧できる。また、双方の計算結果は塩基配列名をキーにしてリンクされている。
成果の活用面・留意点
- 本システムは、他の果樹の発現遺伝子データも処理可能である。
具体的データ
その他
- 研究課題名:カンキツのゲノムインフォマティクス手法の開発
- 課題ID:09-02-07-*-23-04
- 予算区分:交付金・重点強化
- 研究期間:2003~2004年度
- 研究担当者:藤井 浩、清水徳朗、島田武彦、遠藤朋子、大村三男(静岡大)
