カンキツの22K オリゴDNAマイクロアレイの開発
※アーカイブの成果情報は、発表されてから年数が経っており、情報が古くなっております。
同一分野の研究については、なるべく新しい情報を検索ください。
要約
果樹研究所で行った発現遺伝子の解析情報、および、公開情報をもとに設計した 21,495個のカンキツ由来の独立遺伝子を搭載するオリゴDNAマイクロアレイである。搭載されている約60%の遺伝子について遺伝子機能が推定され、データベース化されている。
- キーワード:カンキツ、オリゴアレイ、cDNA、ゲノム、発現遺伝子、データベース
- 担当:果樹研・果樹ゲノム研究チーム
- 連絡先:成果情報のお問い合わせ
- 区分:果樹・育種
- 分類:研究・普及
背景・ねらい
マイクロアレイはガラススライド上に固定された多数の遺伝子の発現を網羅的に解析する技術で、イネやシロイヌナズナなどのモデル植物で強力なゲノム解析ツールとして利用されている。多数の遺伝子の発現情報を同時に解析できるため、遺伝子間の相互作用を捉えたり、大量のクローン中から特定の遺伝子を選抜する遺伝子特定の手法として利用できる。これまでカンキツでは約3,000のcDNAを搭載したcDNAアレイを開発して利用してきたが、搭載遺伝子数の大幅な増加と高い再現性の確保を両立させることが求められてきた。そこで、これまで独自に解析してきた遺伝子情報と、近年多数公開されるようになったオレンジなどの公開配列などの情報を元に、独自のカンキツのオリゴDNAマイクロアレイを開発する。
成果の内容・特徴
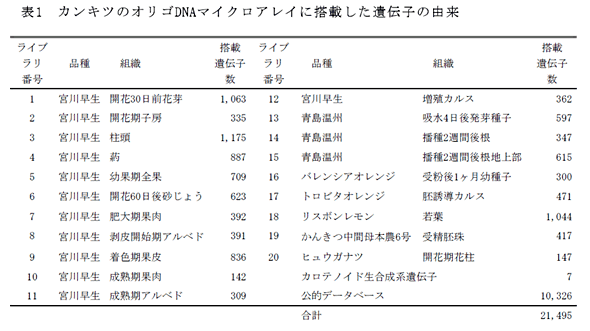
- カンキツの22KオリゴDNAマイクロアレイには、果樹研究所で解析した20のcDNAライブラリ(表1)に由来する約3万の発現遺伝子情報と、公的データベースに公開された約7万の発現遺伝子情報について段階的な配列クラスタリングを行い、最終的に得られた約3万の独立遺伝子配列から選抜された21,495個の遺伝子が搭載されている。
- 本マイクロアレイには内部標準のための検証用プローブが搭載されており、1色法と2色法のいずれにも対応可能である。
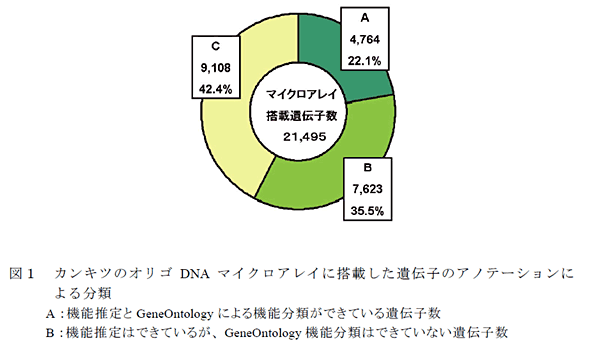
- 公的データベース(GenBank plant, RefSeq plant rna, UniGene A. thaliana, UniGene C. sinensis, UniProt)に対するblastnまたはblastxによる類似性検索(閾値1e-10または1e-30)により、マイクロアレイに搭載した全遺伝子のうちの12,387遺伝子(57.6%)については、機能が推定されている(図1)。このうち、4,764遺伝子(22.1%)についてGeneOntologyにもとづく機能分類が示されている。これらの結果はまとめてデータベース化されており、本マイクロアレイを遺伝子機能の解析に利用することができる。
成果の活用面・留意点
- 本マイクロアレイは、育種分野や植物病理分野などのカンキツのさまざまな生理現象に関連する遺伝子レベルの研究に利用できる。
具体的データ
その他
- 研究課題名:果樹の育種素材開発のための遺伝子の機能解析及びDNA利用技術の開発
- 課題ID:221-j
- 予算区分:二次代謝産物
- 研究期間:2004~2006年度
- 研究担当者:藤井 浩、清水徳朗、島田武彦、遠藤朋子、大村三男(静岡大)
