CAPSマーカーおよびSNPマーカーで構築されるカンキツの標準連鎖地図
要約
本連鎖地図は、カンキツの発現遺伝子の塩基配列をもとに開発したCAPSとSNPなどのDNAマーカーによって構成される。各マーカーの配列などの遺伝子情報によりクレメンティンのゲノムシーケンスと比較でき、カンキツの標準連鎖地図として利用できる。
- キーワード:カンキツ、遺伝子地図、DNAマーカー
- 担当:果樹・茶・果樹ゲノム利用技術
- 代表連絡先:電話 029-838-6453
- 研究所名:果樹研究所・カンキツ研究領域
- 分類:研究成果情報
背景・ねらい
DNAマーカーを用いた実生個体の早期選抜は、播種から開花・結実までに長い期間を要する果樹類において、育種効率を高める極めて有効な手法である。カンキツではこれまでにクレメンティンなどでゲノムシーケンスが解読されているが、我が国のカンキツの育種集団に利用できる有用形質の選抜マーカーを獲得するためには、基盤となる連鎖地図の構築が不可欠である。これまでcDNAライブラリーの大量シーケンスで得た発現遺伝子の塩基配列情報から数多くのDNAマーカーを開発しており、本研究ではカンキツの育種実生集団を用いて標準連鎖地図を作成する。
成果の内容・特徴
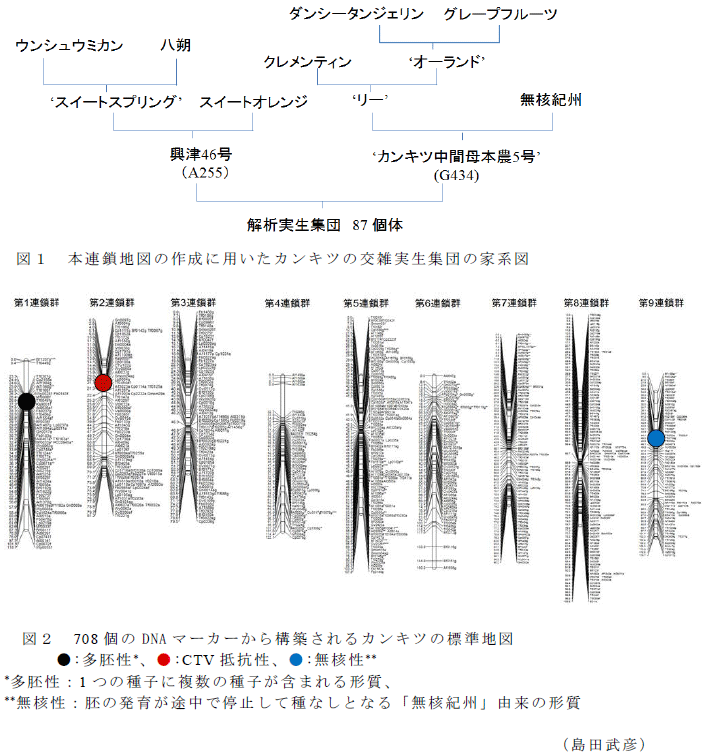
- 本連鎖地図の集団の作成には、カンキツの交雑実生集団(A255×G434)87個体を用いている(図1)。
- 連鎖地図上のDNAマーカーは、593個のCAPS(増幅断片制限断片長多型)、110個のSNP(一塩基多型)、59個のSTS(配列タグ部位)で構成される。76%のDNAマーカーは分析にDNAシーケンサーなどの高額機器を必要としないCAPSマーカーである。地図距離990.9 cM、平均マーカー距離1.4 cMで、連鎖群数はカンキツの染色体基本数n=9と対応する(図2)。
- 連鎖地図上のDNAマーカーは、主要なカンキツ類32品種・系統においてPCRによる増幅を確認しているため、様々なカンキツ類のDNAタイピングに適用できる。
- 708個のDNAマーカーの多くはクレメンティンのゲノムシーケンスver1.0のアノテーション情報を持ち、本連鎖地図とクレメンティンのゲノムシーケンスを容易に比較できる。
- 無核性を制御する遺伝領域は本連鎖地図の第9連鎖群に座乗する。これまでに開発した多胚性とカンキツトリステザウイルス(CTV)抵抗性のDNAマーカーは、第1連鎖群、第2連鎖群にそれぞれ座乗している(図2)。
成果の活用面・留意点
- 本連鎖地図上に位置付けられたDNAマーカーの作成に利用した遺伝子配列は日本DNAデータバンク(DDBJ)に登録しており、遺伝地図の統合、品種識別、親子鑑定などに適用できる。なお、SNPマーカーの分析には、DNAシーケンサーなどの機器が必要である。
具体的データ
その他
- 中課題名:果樹におけるDNAマーカー育種のための高度基盤技術の開発
- 中課題整理番号:142g0
- 予算区分:交付金、委託プロ(ゲノム)
- 研究期間:2004~2014年度
- 研究担当者:島田武彦、藤井浩、遠藤朋子、上田高則、杉山愛子、中野道治、喜多正幸、吉岡照高、清水徳朗、根角博久、生駒吉識、森口卓哉、大村三男
- 発表論文等:Shimada T. et al. (2014) Tree Genetics & Genomes 10:1001-1013
