イネ縞葉枯病抵抗性遺伝子と密接に連鎖する分子マーカー
※アーカイブの成果情報は、発表されてから年数が経っており、情報が古くなっております。
同一分野の研究については、なるべく新しい情報を検索ください。
要約
インド型イネModanに由来するイネ縞葉枯病抵抗性遺伝子(Stv-bi)はイネ第11染色体上腕部に座乗し、分子マーカーであるST10と密接に連鎖している。ST10の塩基配列をもとに設定したST10プライマーによって、PCRで増幅されるDNA断片の有無により、品種・系統個体の抵抗性遺伝子(Stv-bi)の有無を推定できる。
- 担当:北海道農業試験場・生産環境部・ウイルス病研究室
- 担当:同・作物開発部・育種工学研究室
- 担当:愛知県農業総合試験場・普及指導部
- 担当:同・生物工学部・遺伝子工学研究室
- 担当:同・作物研究所・育種研究室
- 連絡先:011-857-9278
- 部会名:生産環境
- 専門:バイテク
- 対象:稲類
- 分類:研究
背景・ねらい
インド型イネModanは、不完全優性であるイネ縞葉枯病抵抗性遺伝子Stv-biを有し、我が国で育成された多くのModan由来の縞葉枯病抵抗性品種は、この遺伝子を持つと考えられている。この遺伝子の遺伝子分子地図上の座乗位置を特定し、近傍の分子マーカーの利用による水稲品種・系統の抵抗性の識別について検討する。
成果の内容・特徴
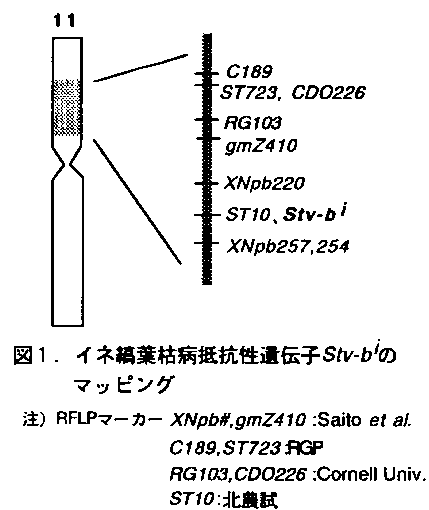
- イネ縞葉枯病抵抗性遺伝子(Stv-bi)はイネ第11染色体上腕部に座乗し、分子マーカーXNpb220とXNpb257(共にSaito、et al.)の間に座乗し、ST10(北農試開発)と密接に連鎖する(0.0cM、図1)。
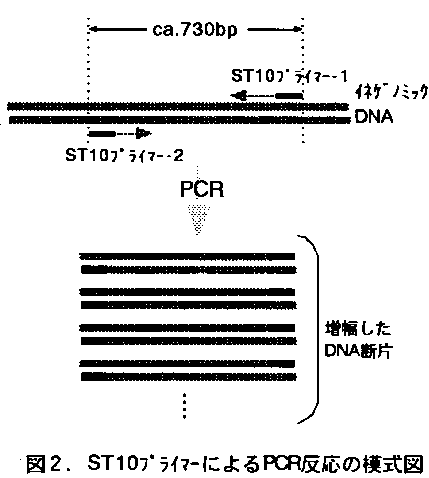
- ST10の塩基配列から設定した、それぞれ20塩基の長さの2種1組のST10プライマーを用い、イネのゲノムDNA(CTAB法により抽出)を鋳型にPCR反応(94°C4分、94°C1分→60°C1分→72°C2分を35サイクル、72°C7分)を行うと、抵抗性品種・系統に特異的な約730bpのDNA断片が増幅する(図2~図3)。
- ST10プライマーを用いたPCR反応で増幅する約730bpのDNA断片の有無によって、イネ縞葉枯病抵抗性遺伝子(Stv-bi)の有無を推定できる。
- Modan以外のインド型イネ(MudgoもしくはIR8)に由来する抵抗性品種においても、この特異的なDNA断片が増幅・検出される。
成果の活用面・留意
- イネ個体の抵抗性遺伝子(Stv-bi)の有無を推定できることから、育種現場における間接選抜への利用が可能。
- 鋳型となるイネゲノムDNAの精製度に応じて、PCR反応条件(特に、アニーリング温度やサイクル数)を考慮する必要がある。
- PCR反応にはCTAB法により抽出した抵抗性・感受性の標準品種のDNA試料を同時に供試するのが望ましい。
- 本成果は北農試と愛知県農総試との共同研究によって得られたものであり、特許出願を検討中である。
具体的データ

その他
- 研究課題名: イネ縞葉枯病抵抗性遺伝子の解析
- 予算:バイテク先端技術開発(イネ・ゲノム)
- 研究期間:平成8年度(平成3~9年)
- 発表論文等:グラフィカルジェノタイプによるイネ縞葉枯病抵抗性品種の分類、育雑、46(別1)、1996
