アガロースゲルで検出できるSSRマーカーを用いたアカクローバの高密度連鎖地図
※アーカイブの成果情報は、発表されてから年数が経っており、情報が古くなっております。
同一分野の研究については、なるべく新しい情報を検索ください。
要約
アカクローバの高密度連鎖地図をSSRおよびRFLPマーカーを用いて作成した。SSRマーカーはアガロースゲルにより簡易・安全に検出できる。また、マーカーは遺伝子領域に由来しているため、他マメ科植物に対する応用性が高い。
- キーワード:アカクローバ、高密度連鎖地図、SSRマーカー、アガロースゲル
- 担当:北海道農研・作物開発部・マメ科牧草育種研究室
- 連絡先:電話011-857-9272、電子メールsisobe@affrc.go.jp
- 区分:北海道農業・作物、畜産草地・飼料作物、作物・生物工学
- 分類:科学・普及
背景・ねらい
高密度連鎖地図は収量、早晩性など農業形質に関与する量的形質遺伝子座(QTL)の検出精度を高め、選抜効果の高いDNAマーカーを開発するうえで非常に有用である。SSR(Simple Sequence Repeats)マーカーは共優性かつPCRベースで解析できるため、多くの植物種で開発が行われている。一方、これらのマーカーは数bpの多型に基づいて検出されるため、多型検出に煩雑な手法を用いる場合が多い。そこで、簡易・安全なアガロースゲルで検出ができるSSRマーカーを用いた高密度連鎖地図を作成する。
成果の内容・特徴
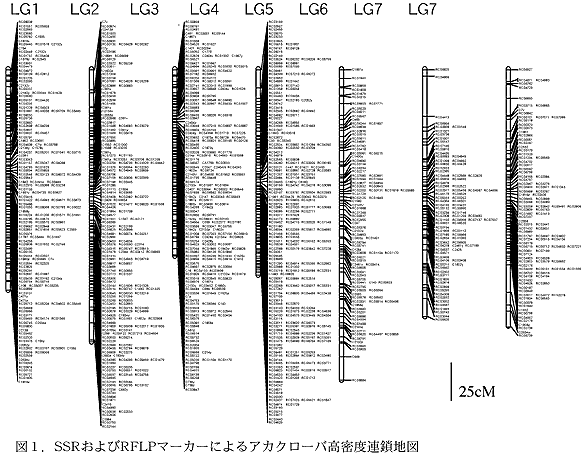
- 本連鎖地図は1157マーカーからなり、アガロースゲルで検出できるSSRマーカーを用いた高密度連鎖地図としては唯一のものである(図1,図2)。
- マーカーの内訳はSSRマーカーが1013、RFLPマーカーが144で、連鎖地図の全長は989cM、マーカー間の平均距離は0.9cMである(図1)。
- マッピング集団「HRxR130」は日本品種とスイス品種のF1由来のクローン「HR」とロシア品種由来のクローン「R130」によるF1188個体である。
- SSRマーカー、RFLPマーカーはcDNAライブラリー等を用いて開発し、ともにアカクローバの遺伝子領域に由来している。
- SSRマーカーの70%がシロクローバのDNAでアカクローバとほぼ同じサイズのバンドを検出した。同様に26%がマメ科モデル植物Medicago truncatulaで、21%がエンドウでアカクローバとほぼ同サイズのバンドを検出しており、本SSRマーカーは他マメ科植物に対する応用性が高い(表1)。
成果の活用面・留意点
- 本連鎖地図は高密度なため、精度の高いQTL解析を行うことができ、選抜効果の高いDNAマーカー開発に活用できる。
- 本成果は(財)かずさDNA研究所との共同研究によるものであり、連鎖地図上のSSRマーカープライマー情報およびRFLPシーケンス情報を論文およびwebサイト(http://www.kazusa.or.jp/)にて全て公開する。
- 連鎖地図上のSSRマーカーは全てアガロースゲルを用いて簡易に多型検出ができる。
- 連鎖地図上のマーカーはシンテニー解析や候補遺伝子解析およびマメ科植物のアンカーマーカーに活用できる。
具体的データ


その他
- 研究課題名:マメ科牧草におけるDNAマーカーの効率的作出法の開発及び遺伝地図作成
- 課題ID:04-08-02-(01)-10-04
- 予算区分:DNAマーカー
- 研究期間:2003~2004年度
- 研究担当者:磯部祥子、奥村健治、イリーナ・クリメンコ(ウィリアムス飼料作研究所)、
ニコライ・コズロフ(ウィリアムス飼料作研究所) - 発表論文等:Isobe et al. (2003) Theor Appl Genet 108:105-112
