SSRマーカーに基づくダイズ統合連鎖地図
※アーカイブの成果情報は、発表されてから年数が経っており、情報が古くなっております。
同一分野の研究については、なるべく新しい情報を検索ください。
要約
3種の組換え自殖系統群を用いて構築したダイズの統合分子連鎖地図は、SSRマーカーを中心とする1811のDNAマーカーからなり、各マーカーの多型情報を提供する。
- キーワード:ダイズ、SSRマーカー、組換え自殖系統、連鎖地図、マーカー育種
- 担当:北海道農研・低温耐性研究チーム、九沖農研・大豆育種研究九州サブチーム、近中四農研・大豆育種研究近中四サブチーム
- 代表連絡先:電話011-857-9260、電子メールseika-narch@naro.affrc.go.jp
- 区分:北海道農業・生物工学、作物
- 分類:研究・普及
背景・ねらい
農業上重要な形質の解析と育種的利用において、正確で効率的な遺伝子型の判別を可能にするDNAマーカーの果たす役割は非常に大きい。なかでも、SSR(Simple Sequence Repeats)マーカーは共優性でかつまたPCRにより簡便に解析できるため、有用性が高い。ダイズにおいても多数のSSRマーカーが開発されてきたが、ゲノムサイズが比較的大きいためその数はまだ十分ではない。そこで、育種現場での利用を前提に、簡易なゲル電気泳動で識別可能なSSRマーカーを用いて、複数の組換え自殖系統について個別に連鎖地図を作成し、位置情報を統合することにより高密度連鎖地図を構築する。また、主要品種における各マーカーの遺伝子型を調査する。
成果の内容・特徴
- 統合連鎖地図は、SSRマーカーを中心に1811のPCRマーカーからなり、全長2422.9cM、マーカー間の平均距離は1.36cMである(図1)。
- マーカーの内訳は、アメリカ農務省のSSRマーカーが596、かずさDNA研究所が開発したEST-SSRマーカーが1,074、そして、千葉大学が開発したSSRとSTSマーカーが141である(図1)。このうち、468が新しく位置づけられたマーカーである。
- 10cM以上にわたってマーカーが存在しない領域が7カ所ある。とくに、連鎖群C1(第4染色体)のSat_235とGMES1325の間には大きなマーカーの欠落部が存在する(図1、赤色の縦線部分)。
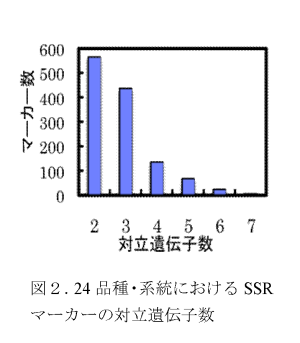
- 国内主要品種を含む24品種・系統についてアガロースゲル電気泳動のような簡便な方法で多型を検出した場合、各マーカーの対立遺伝子数は2が最も多く、最大7を示す(図2)。
成果の活用面・留意点
- 3つの連鎖地図を統合して作成した地図であるため、細部のマーカー順序は実際とは異なる場合がある。ゲノム情報や連鎖解析の結果から確認する必要がある。
- マーカー情報は下記のホームページからダウンロード可能である。http://dnaresearch.oxfordjournals.org/cgi/content/full/dsp010/DC1
具体的データ

その他
- 研究課題名:作物の低温耐性等を高める代謝物質の機能解明とDNAマーカーを利用した育種素材の開発
- 中課題整理番号:221e
- 予算区分:委託プロ(新農業展開)
- 研究期間:2007~2009年度
- 研究担当者:石本政男、黄太暎、佐山貴司、高橋将一、高田吉丈、中本有美、船附秀行、久野裕(かずさDNA研)、笹本茂美(かずさDNA研)、佐藤修正(かずさDNA研)、田畑哲之(かずさDNA研)、河野いづみ(STAFF研究所)、星雅子(千葉大園芸)、塙正義(千葉大園芸)、矢野千鶴(千葉大園芸)、夏正俊(生物研)、原田久也(生物研)、喜多村啓介(北大院農)
- 発表論文等:1) Hwang T.-.Y. et al. (2009) DNA Research 16:213-225
