イネいもち病抵抗性遺伝子と連鎖する分子マーカー
※アーカイブの成果情報は、発表されてから年数が経っており、情報が古くなっております。
同一分野の研究については、なるべく新しい情報を検索ください。
要約
イネいもち病抵抗性遺伝子(Pii、Pik-m、Pit)と分子マーカーの間で連鎖分析を行い、各いもち病抵抗性遺伝子と連鎖する分子マーカーを見出した。
- 担当:九州農業試験場・水田利用部・稲育種研究室
- 連絡先:0942-52-5055-(310)
- 部会名:総合農業(作物生産)、水田作
- 専門:バイテク
- 対象:稲類
- 分類:研究
背景・ねらい
いもち病抵抗性遺伝子(Pii、Pik-m、Pit)とRFLPマーカー等の分子マーカーとの連鎖関係を明らかにし、分子マーカーを慣行育種における抵抗性品種育成のマーカーとして利用するとともに、抵抗性遺伝子クローニングの素材を提供する。
成果の内容・特徴
- コシヒカリ/石狩白毛のF3系統を用いてPiiと連鎖するDNA断片を増幅するPCRプライマーを探索し、プライマ-CA05を見出した。Piiはプライマ-CA05を用いたPCR反応によって増幅するDNA断片の有無と5.9cMの遺伝距離で連鎖する。このDNA断片はPiiを持つ品種、「石狩白毛」、「キヌヒカリ」、「ヒノヒカリ」のいずれにおいても増幅する(図1)。
- コシヒカリ/ツユアケのF3系統を用いてRFLPマーカーとPik-mの連鎖分析を行った結果、Pik-mは第11染色体上に座乗することが明らかになった。Pik-mはRFLPマーカ-R1506と密接に連鎖する(0.0cM)。
- コシヒカリ/K59(Pitの代表品種)のF3系統を用いて、RFLPマーカーとPitの連鎖分析を行った結果、Pitは第1染色体上に座乗することが明らかになった。PitはCA59プライマーを用いたPCR反応によって増幅するDNA断片の有無と密接に連鎖する(0.0cM、図2)。
成果の活用面・留意点
- 本研究で見出した分子マーカーを用いることで、イネいもち病抵抗性遺伝子(Pii、Pik-m、Pit)を持つ個体、系統を間接的に選抜できる。
- 分子マーカーを用いていもち病抵抗性を選抜する際には、あらかじめ両親品種での多型の出現を確認しておく必要がある。
具体的データ

図1 CA05プライマーによって増幅するPiiを持つ品種に特異的なDNA断片(矢印)
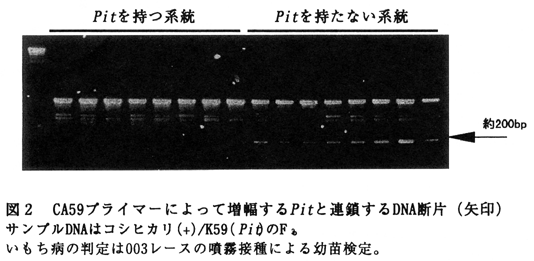
図2 CA59プライマーによって増幅するPitと連鎖するDNA断片(矢印)
その他
- 研究課題名:いもち病・白葉枯病抵抗性遺伝子の解析
- 予算区分 :バイテク先端技術開発(イネ・ゲノム)、経常
- 研究期間 :平成10年度(平成6~9年)
