遺伝子構造に基づいた土壌細菌群集解析手法
※アーカイブの成果情報は、発表されてから年数が経っており、情報が古くなっております。
同一分野の研究については、なるべく新しい情報を検索ください。
要約
土壌から分離した細菌の16S rDNAの制限酵素切断片をデンシトグラムで数値情報化し、既知細菌の塩基配列情報から構築したデータベースからの検索による分類群の推定を行うことで、多岐に渡る土壌細菌群集の構成を簡易に解析することができる。
- 担当:九州農業試験場・生産環境部・土壌微生物研究室、地域基盤研究部・病害遺伝子研究室
- 連絡先:096-242-1150
- 部会名:農業環境(農業生態)・生産環境
- 専門:土壌
- 分類:研究
背景・ねらい
農耕地土壌は最も微生物の多様性に富む環境の一つであり、生態解明においては多岐に渡る細菌の分類が必要とされる。近年16S rDNAの塩基配列が細菌の系統分類において最も信頼できる指標として確立したことにより、制限酵素切断パターンが分類体系を反映した簡易な分類指標となる可能性がある。ここでは、データベースに登録してある16S rDNAの塩基配列を利用した土壌細菌群集解析に適応可能な分類群の推定手法の開発を行う。
成果の内容・特徴
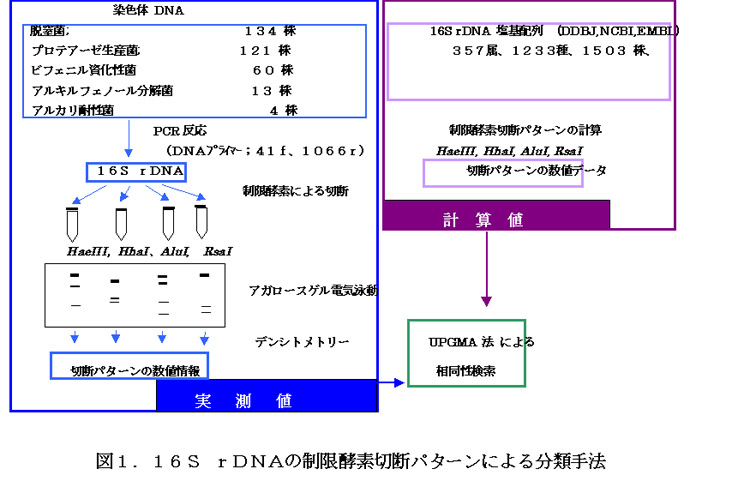
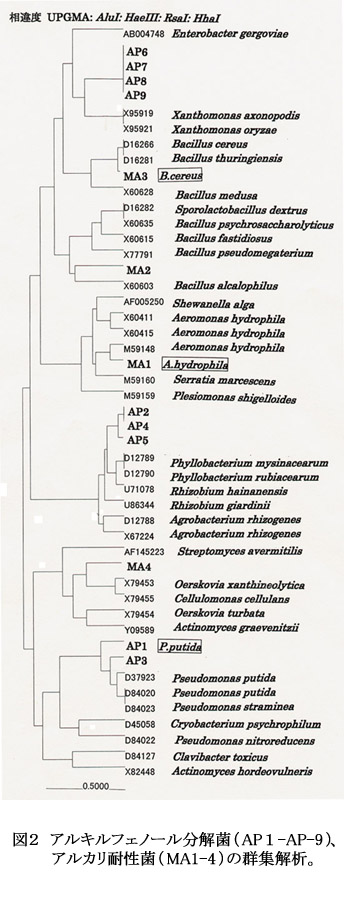
- データベース(DDBJ,NCBI,EMBL等)に登録してある1503株の細菌(357属、1233種)の16S rDNAの塩基配列から、4種類の制限酵素(HaeIII, Hha I, Alu I, Rsa I)による切断長を計算し計算値データベースとし、分離菌株由来の染色体DNAを鋳型として、ユニバーサルプライマー(41f, 1066r)を用い16S rDNAを増幅後、制限酵素で切断し、電気泳動後デンシトグラムで断片長を測定し実測値データベースとする(図1)。UPGMA法(非加重結合法)により実測値と相違度が低い計算値を有する菌を検索し、グループ化及び各グループの分類群の推定を行うソフトウェアーを作成した(図2)。
- 家畜スラリー投与飼料作畑土壌から分離した脱窒菌(134株)は本法で13グループに分類され、その代表株(32株)の16S rDNA部分塩基配列に基づく相同性検索による同定結果は、本法による同定結果と一致する(表1)。
- プロテアーゼ生産菌(121株)の生態解明では、通常の土壌(水田、畑)ではBacillus属細菌が、家畜スラリーを過剰投与し土壌プロテアーゼ活性が高い飼料作畑土壌ではSerratia marcescens, Xanthomonas属、Pseudomonas属細菌等のグラム陰性細菌が主要な土壌プロテアーゼの供給源となっている可能性が示唆された。
成果の活用面・留意点
- この手法は、16S rDNAの塩基配列の相違を反映した多岐に渡る分離細菌の群集解析手法として適応可能である。
- この手法はrDNAの構造が異なる古細菌には適応できない。また同定法としての精度はデータベースに登録している細菌の種類・数に依存し、現状ではEnterobacteriaceaeではデータ数が少なく正確には種名が推定できない。
具体的データ

その他
- 研究課題名:土壌の物質代謝に関与するグラム陰性細菌の簡易同定手法の開発、有機塩素系化合物の分解に関与する土壌微生物の生態と分解能の判定、バイオマス窒素代謝関連微生物活性の動態
- 予算区分:経常研究、フロンテイア研究(環境修復)、一般別枠研究(物質循環)
- 研究期間:平成11年度(平成10年~12年、平成8年~10年、平成4年~10年)研究担当者:渡邊 克二・奥田充・古賀伸久(現北海道農業試験場・生産技術チーム)
