イネいもち病菌の非病原性遺伝子Avr-PiksおよびAvr-Pishの特異的検出法
※アーカイブの成果情報は、発表されてから年数が経っており、情報が古くなっております。
同一分野の研究については、なるべく新しい情報を検索ください。
要約
イネいもち病菌の非病原性遺伝子Avr-PiksおよびAvr-Pishは、それぞれプライマ-PksF3とPksR1およびPshF4とPshR4の組み合せを用いたPCR法によって検出できる。
- 担当:九州農業試験場・地域基盤研究部・病害生態制御研究室
(九州沖縄農業研究センター地域基盤研究部病害生態制御研究室) - 連絡先:096-242-7729
- 部会名:総合農業(生産環境)、病害虫
- 専門:作物病害
- 対象:稲類
- 分類:研究
背景・ねらい
いもち病菌の病原性レースは、接種した菌株に対するイネ苗の反応型によって検定されている。しかし、病斑型は苗の生育状態や環境条件によって変動することがあるため、判定に熟練を要することが多く、簡易検定法の開発が望まれている。そこで、既に明らかな高密度分子連鎖地図上の非病原性遺伝子に近いマーカーをSTS(Sequence Tagged Site)化し、レースの簡易推定法における有効性を明らかにする。
成果の内容・特徴
- Avr-PiksおよびAvr-Pishに強く連鎖するマーカ-H16/1200、E15/2500をクローニングし、その塩基配列から、それぞれの非病原性遺伝子を安定に検出するためのプライマーセットとPCRの条件を決定した(図1)。
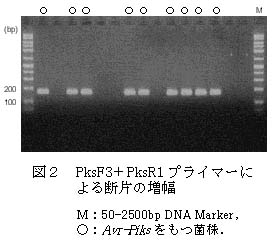
- Avr-Piksをもつ菌株では、プライマ-PksF3とPksR1を用いたPCRにより、約170bpの増幅断片が検出される(図2)。
- Avr-Piksをもたない菌株では、断片の増幅が認められない(図2)。
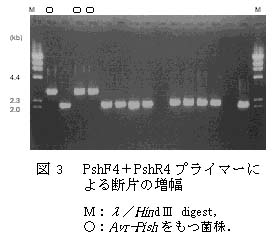
- Avr-Pishをもつ菌株では、プライマ-PshF4とPshR4を用いたPCRにより、約2700bpの増幅断片が検出される(図3)。
- Avr-Pishをもたない菌株では、約2000bpの断片が増幅されるか、あるいは断片の増幅が認められない(図3)。
- 以上より、上記の2通りのプライマーの組み合せにより、いもち病菌の非病原性遺伝子Avr-PiksおよびAvr-Pishの特異的検出が可能である。
成果の活用面・留意点
- いもち病菌レースの簡易推定法の端緒となる。
- STS化されたマーカーは、両非病原性遺伝子の単離にも極めて有効である。
- レース判別品種が異なっている各国産いもち病菌株の病原性の比較が可能になる。
具体的データ

その他
- 研究課題名:病原性変異関与遺伝子の同定と病原性遺伝様式の解明(九州農試)
- 予算区分:作物対応研究(次世代稲作)
- 研究期間:平成12年度(平成10?12年)
