イネいもち病菌の動態を個体レベルで追跡するためのDNAマーカー
※アーカイブの成果情報は、発表されてから年数が経っており、情報が古くなっております。
同一分野の研究については、なるべく新しい情報を検索ください。
要約
イネいもち病菌菌株を特定するDNAマーカーは、STS化することにより一般的PCR法で安定に増幅されるため、いもち病菌動態の個体レベルの追跡に有効である。この際、鋳型DNAは、菌体をセルラーゼで処理するだけで効率的に調製できる。
- キーワード:イネいもち病菌、分子マーカー、STS、個体群動態
- 担当:九州沖縄農研・地域基盤研究部・病害生態制御研究室
- 連絡先:096-242-7729
- 区分:九州沖縄農業・病害虫
- 分類:科学・普及
背景・ねらい
イネの最重要病害であるいもち病菌において、個々の菌株の伝染過程や個体群としての動態については不明な点が多い。そこで、いもち病菌の動態を個体レベルで追跡するために、菌株に特異的なDNAマーカーのSTS化およびその効率的検出法を開発する。
成果の内容・特徴
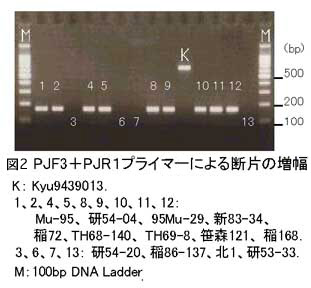
- ともに21塩基からなるプライマーPJF3とPJR1 の組合せをもちいたPCR法(図1)によって増幅される約650bpの断片は、供試した500以上の菌株の中でKyu9439013株にのみ検出される(図2)。その他の菌株では、約165bpの断片が増幅されるか、断片の増幅が認められない(図2)。
- このDNAマーカーは、Kyu9439013株をイネ上で7回、培地上で50回継代した場合にも安定に検出される。
- DNAマーカーをSTS化した後は、一般的なPCR法の増幅産物を電気泳動することによって目標菌株を識別できるため、従来のAFLP法やRAPD法における煩雑性あるいは再現性の問題が解決される。さらに、一反応に要するDNAポリメラーゼは0.5ユニットで十分であり、例えば同量の反応液によるPot2 rep-PCR法に較べて5倍も経済的である。
- このDNAマーカーを用いると、他の菌株が混在する場合にも目標菌株を確認できる。従って、検定のための菌体は単胞子分離を必要としない。
- PCR反応に供試する鋳型DNAの調製法は、基本的には、少量の培養菌体を1%(w/v)セルラーゼを含む50mM Tris-HCl(pH7.4)、10mM EDTA液に懸濁し、37 °で90分間保つだけの操作であり、多数のサンプルを対象とする場合には極めて有効である(図3)。
- 得られた鋳型DNA液は、4 °・2週間の保存でも安定であり、余裕をもった実験設計が可能である。
成果の活用面・留意点
- Kyu9439013株以外のいもち病菌株、あるいは他の病原微生物についても、特異的なDNAマーカーをSTS化することにより、それらの生態解明に応用可能である。
具体的データ


その他
- 研究課題名:九州・沖縄地域におけるイネいもち病菌系統の動態と変異性の解明
- 予算区分:交付金
- 研究期間:1999~2001年度
- 研究担当者:平八重一之、中島 隆、藤田佳克(九州研)、山口純一郎(佐賀農業セ)
- 発表論文等:(1)イネいもち病菌Kyu9439013株を特定する分子マーカー、九州病害虫研究会報、47巻、2001。
(2)自家採種を繰り返している圃場におけるイネいもち病菌の個体群動態、
第64回九州農業研究発表会専門部会発表要旨集、2001。
