マイクロチップ電気泳動法を利用した微生物遺伝子の類縁性解析システム
※アーカイブの成果情報は、発表されてから年数が経っており、情報が古くなっております。
同一分野の研究については、なるべく新しい情報を検索ください。
要約
塩基配列ファイルから様々な遺伝子の制限酵素切断長データへ変換し実測値切断長データと比較可能なシステムを開発し、マイクロチップ電気泳動装置から制限酵素切断長データを取り込むことで短時間に高精度で様々な遺伝子の類縁性の解析が行なえる。
- キーワード:マイクロチップ電気泳動装置、制限酵素切断長データ、類縁性解析システム
- 担当:九沖農研・環境資源研究部・土壌微生物研究室
- 連絡先:電話096-242-1150、電子メールkatsuji@affrc.go.jp
- 区分:九州沖縄農業・生産環境(土壌肥料)
- 分類:技術・参考
背景・ねらい
遺伝子の類縁性検索は主に塩基配列の決定により行われてきたが、分析に関する機器や試薬類が国外特許で占められていることや、煩雑な操作を必要とすることから研究用技術に留まっている。よってここでは簡易な分析方法として普及可能な遺伝子の類縁性検索システムを開発する。
成果の内容・特徴
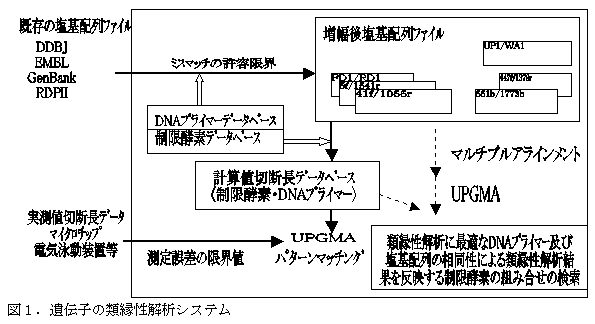
- 本システムは既存の塩基配列データからプライマー毎、制限酵素毎に計算値切断パターンをデータベース化し、電気泳動装置から得た制限酵素切断パターンの実測値から様々な種類の遺伝子の類縁性が解析可能である(図1)。
- 本システムにより16S rDNA遺伝子の41f/1066rプライマーに対し576属の細菌からなる4370種類のデータベースを作成しており、様々な種類の細菌の分類が可能である。16S rDNA遺伝子のRD1/FD1や8F/1541R等の他のプライマー毎にデータベースがあり様々な領域の遺伝子解析に有効である。
- 本システムにより塩基配列の決定による類縁性解析結果を反映するDNAプライマー、制限酵素の組み合せが予測、検索可能であり(図1)。GyraseB遺伝子やN2O還元酵素遺伝子(nosZ)等他の遺伝子でもプライマー毎にデータベース化され、HaeIIIやHhaIの制限酵素切断パターンから遺伝子の類縁性解析が可能である。
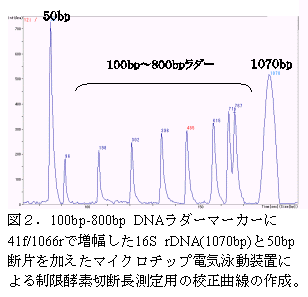
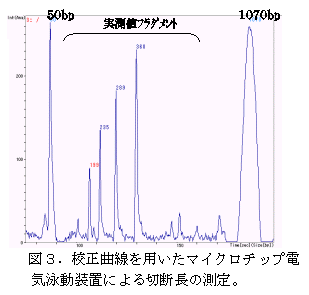
- 切断前の増幅遺伝子を校正曲線用マーカーとして使用することで(図2)、マイクロチップ電気泳動装置で高精度・短時間(誤差精度4から7%、12サンプル/6分;大型アガロースゲル、誤差精度10%、ゲル作成から鎖長測定まで15から20サンプル3時間から4時間)で切断長測定が可能であり(図3)、本システムで多くの場合実測値と一致する計算値切断パターンを持つ遺伝子が検索され、短時間に多数の遺伝子の類縁性解析が精度良く行える。
成果の活用面・留意点
- 塩基配列の決定法と比べて、ランニングコストが安く、操作が簡便であり、多数のサンプルの分析が短時間で終了することから分析のルーチン化に適している。
- 計算値切断パターンと実測値切断パターンが一致しない場合は5塩基差以内の異なる切断片間の分離が出来ない事が原因であり、キャリブレーションプログラムを開発する必要がある。
- 塩基配列の決定法と比べて、異なる複数の遺伝子(異なる微生物の混合物;16S rDNAや18S rDNA等の多コピー性遺伝子で異なる配列を持つ遺伝子)を同時に解析可能であるが、同じ長さの切断片を生じる遺伝子群の解析には別途解析アルゴリズムの開発が必要である。
具体的データ
その他
- 研究課題名:遺伝子の制限酵素切断長多型を利用した効率的な微生物の類縁性解析手法の開発、
内分泌かく乱物質の環境での脱ハロゲン化分解 - 予算区分:運営費交付金・環境ホルモン
- 研究期間:2000~2003年度
- 研究担当者:渡邊克二・奥田充・吉川博道(九州共立大学)
- 発表論文等:1)渡邊・奥田 (2000) 遺伝子の類縁性解析方法及び遺伝子の類縁性解析システム.特願2000-215134
2)Watanabe and Okuda (2001) UP 98:2345-2356.
