イグサ品種「ひのみどり」を識別するSSRマーカー
※アーカイブの成果情報は、発表されてから年数が経っており、情報が古くなっております。
同一分野の研究については、なるべく新しい情報を検索ください。
要約
単純反復配列(SSR)を利用して熊本県育成のイグサ品種「ひのみどり」を識別する技術を開発した。畳表から抽出したDNAを鋳型にマルチプレックス PCRにより100から250塩基対のDNA断片を増幅することで,高精度かつ安定にイグサ品種「ひのみどり」を識別可能である。
- キーワード:イグサ、畳表、品種識別、DNAマーカー、SSR
- 担当:九州沖縄農研・作物機能研究部・育種工学研究室
- 連絡先:電話 096-242-7564、電子メール cyto1952@affrc.go.jp
- 区分:九州沖縄農業・植物バイテク
- 分類:行政・普及
背景・ねらい
平成15年の改正関税定率法の施行により育成者権侵害物品の輸入差し止めが可能になり、その技術的裏付けとしてイグサの品種識別技術の確立が求められてい る。従来の技術は高温で乾燥されたイグサ製品の断片化したDNAに対して安定な結果が得られないため、畳表からのDNA抽出技術の確立と安定で確実な品種 識別技術を確立する。
成果の内容・特徴
- 劇薬を使用しない改変CTAB法により、畳表のイグサの先端部から切り取った長さ5 cm程度の試料から4 μg程度のDNAを簡易に抽出できる。
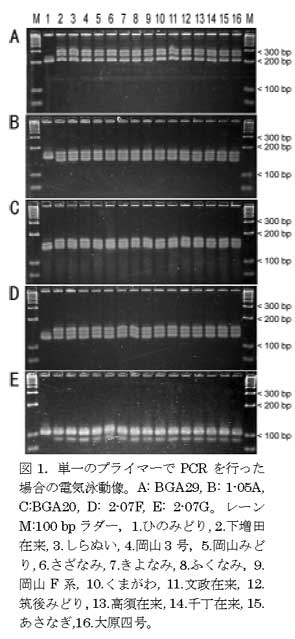
- 5種類の単純反復配列(SSR)領域を利用して品種識別するDNAマーカーを開発した。PCRのサイクルは,96℃ 2分の変性の後,(96℃ 5秒,58℃ 15秒,72℃ 30秒)を30回繰り返し,最後に72℃ 2分間の伸長反応を行う。4%アガロースゲル電気泳動で増幅産物を分離すると,品種「ひのみどり」では1-2本、その他の16品種では2-3本のバンドが 観察され品種の識別が可能である(図1)。
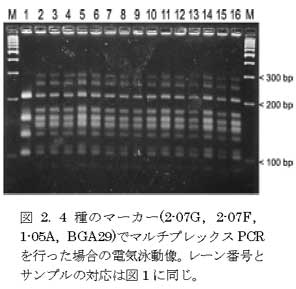
- 開発したSSRマーカーのうち4種類を併用するマルチプレックスPCR法により複数の遺伝子座の遺伝子型を同時に検定できるため、確実な識別が可能である(図2)。
成果の活用面・留意点
- 開発された5種類のマーカーはいずれも「ひのみどり」にはバンドが見られないことが特徴である。従って,製品の検定にあたっては乾燥茎1本ごとにDNAの抽出とPCRを行う必要がある。
- DNA抽出、プライマーの塩基配列、遺伝子型の検定に至る一連の具体的な手法は農林水産省種苗課のホームページで分析マニュアルとして公開されている(http://www.hinsyu.maff.go.jp/DNAgaido/san4.pdf)。
- DNA抽出からPCRまでに必要な試薬をキット化して市販する予定である。
具体的データ
その他
- 研究課題名:SNP分析によるイグサ品種識別法の開発
- 課題ID:07-07-01-**-09-03
- 予算区分:ネギ・イグサ
- 研究期間:2001~2003年度
- 研究担当者:土門英司、斎藤彰、木村貴志
- 発表論文等:
1) 農林水産省生産局種苗課、植物のDNA品種識別についての基本的留意事項より、分析マニュアル”DNA分析によるいぐさ品種「ひのみどり」の識別
2) 特願2003-408328 「SSR法によるイグサ品種識別」
3)SSR分析によるイグサ品種識別法の開発、土門英司・木村貴志・斎藤彰 (2003.9) 育種学研究5、別冊2号:178
