ソラマメウイルトウイルス弱毒株(34B)の遺伝子検出法
※アーカイブの成果情報は、発表されてから年数が経っており、情報が古くなっております。
同一分野の研究については、なるべく新しい情報を検索ください。
要約
わい性リンドウのソラマメウイルトウイルスによるモザイク病防除のための本ウイルス弱毒株はRT-PCR後のDNA断片の制限酵素消化によって強毒株と識別することができる。
- キーワード:ソラマメウイルトウイルス、リンドウ、弱毒ウイルス、塩基配列、遺伝子増幅、制限酵素消化、系統識別
- 担当:中央農研・病害防除部・ウイルス病害研究室、病害防除システム研究室
- 連絡先:0298-38-8932
- 区分:共通基盤・病害虫
- 分類:科学・参考
背景・ねらい
埼玉県における鉢物用わい性リンドウにモザイク症状を生じさせるウイルスの一つにソラマメウイルトウイルス(Broad bean wilt virus: BBWV)が報告されている。本病の有効な防除法の一つとして弱毒ウイルスの開発が進められ、いくつかの有望株が得られるにいたった。そこで、弱毒ウイル スの安定的・効率的な利用法を検討するため、リンドウから弱毒ウイルスを容易に検出できる技術の開発を図る。
成果の内容・特徴
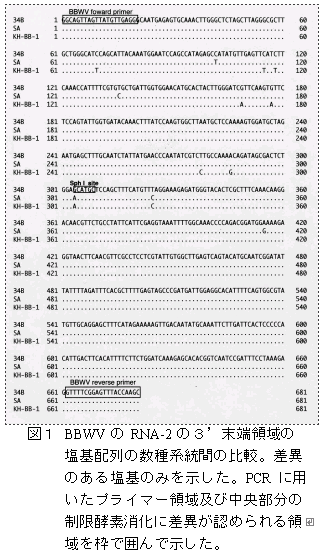
- BBWVの弱毒株(34B)及び強毒株(SA、KH-BB-1)のRNA-2について、3' 側より 約2300 塩基を決定した。
- 各分離株の塩基配列の相同性は極めて高かったが、わずかな違いも認められた。
- 弱毒株と強毒株の塩基配列の違いが認められたもので、制限酵素による消化に差異が生じる可能性があるものを解析した結果、図1の304番目に弱毒株では制限酵素Sph I によって切断されるが、強毒株では切断されない塩基の変異が見出された。
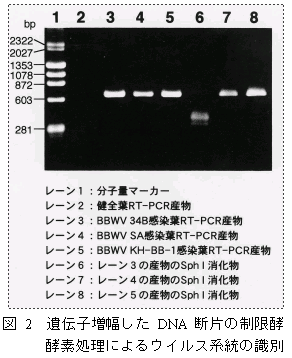
- 設計したプライマーを用いて遺伝子増幅を行ったところ、期待される681塩基に近いサイズのDNA断片が得られた(図2、レーン3, 4, 5)。
- これらを制限酵素Sph I で処理し、電気泳動したところ、弱毒株は消化され短い断片になったが、強毒株は消化されなかった(図2、レーン6, 7, 8)。
- 以上の結果から本検出法は病原性を異にするウイルス系統の簡易識別及びこれらウイルス系統の自然環境下での動態モニター等に利用可能である。
成果の活用面・留意点
- 現地栽培リンドウでの弱毒ウイルスの定着等、ウイルスの動態を解析するのに必要な基礎技術として用いられる。
具体的データ
その他
- 研究課題名:リンドウの弱毒ウイルスの検出および定量技術の改良
- 予算区分:地域先端
- 研究期間:1999~2001年度
- 研究担当者:小林有紀(北農セ)、宇賀博之(埼玉農総研)、庄司俊彦(埼玉農総研)、萩原恭二(特別研究員)、本田要八郎、大村敏博
- 発表論文等:小林ら (2001) 日本植物学会報 第67巻(2号):173-174
