米の品種識別用SNP/STSマーカー
※アーカイブの成果情報は、発表されてから年数が経っており、情報が古くなっております。
同一分野の研究については、なるべく新しい情報を検索ください。
要約
日本の栽培稲品種間で見い出されるゲノムの多型領域の多くは、2種類の遺伝子型で構成されている。このゲノム情報を活用して開発したSNP判別マーカーとSTSマーカーを用いることで、米1粒の品種識別を簡易で安定的かつ短時間に行うことができる。
- キーワード:SNP、STS、ゲノム情報、米1粒、品種識別
- 担当:中央農研・北陸地域基盤研究部・稲遺伝解析研究室
- 連絡先:電話025-526-8251、電子メールhtabuchi@affrc.go.jp
- 区分:関東東海北陸農業・北陸・生物工学
- 分類:科学・普及
背景・ねらい
改正JAS法の施行により米の品種名の表示が義務づけられ、また混米による不正を防止する必要性等から、簡易で精度の高い米の品種識別技術の開発が求められている。そのため、まず日本における栽培稲の品種間で特徴的に異なるゲノム領域を見い出し、塩基配列を解析する。次にここで得られた情報を利用することで、少数のマーカーで多数の品種の識別を可能にするモデルを提唱する。また、SNP(1塩基多型)を利用した新規タイプのマーカーを作成し、従来型のSTS(塩基配列標識部位)マーカーと同様に安定的な米1粒の品種識別を短時間に行うことが可能であることを実証する。
成果の内容・特徴
- 在来種を含む日本の栽培稲のDNAを用い、AFLP(増幅制限酵素断片長多型)解析で同定される複数の多型領域の塩基配列を決定し比較すると、2種類の遺伝子型に分けられる(図1)。この情報を利用することで、品種を2グループに大別できる。それぞれのグループにどの品種が分類されるかは、多型領域ごとに異なる。
- 各々の多型領域を構成する2種類の遺伝子型のDNA塩基配列を識別可能な、SNP判別マーカーまたはSTSマーカーを作成する。これらを適宜組み合わせて使用することで、AFLP未解析の品種も含め多数の品種の識別を可能にするモデル例を、図2に示す。
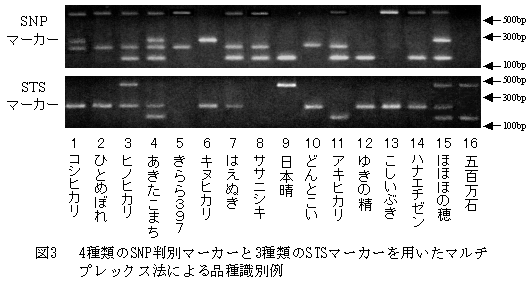
- 全国作付シェア2%以上の7品種、その他の品種および北陸4県でシェアの高い5品種の合計16品種を用い、上記マーカーおよびモデルの実証試験を行った。精米または玄米1粒から簡易法により粗DNAを調製し、4種類のSNP判別マーカーと3種類のSTSマーカーを用いたマルチプレックスPCRを2回行い、増幅バンドパターンを比較することにより、簡易で安定的な品種識別が可能である(図3)。なお、DNA調製から結果が得られるまでの所要時間は約4時間である。
- 遺伝的に近い品種を識別する場合、複数塩基が連続して欠損または変異していないと作成できないSTSマーカーと比較し、1塩基のゲノムの違いを利用するSNP判別マーカーを用いることで、より効率的なマーカー設定が可能である。
成果の活用面・留意点
- 図1、図3に示していない品種については未調査であるが、ゲノム情報を利用しマーカー数を増加させることにより、より多くの品種識別が可能となる。
- 本成果内容の一部は、特許出願中である。
具体的データ


その他
- 研究課題名:ゲノム研究手法・資源を活用した水稲の新規な遺伝・育種研究法の開発
- 予算区分:交付金
- 研究期間:2002年度
- 研究担当者:田淵宏朗、林敬子、芦川育夫
- 発表論文等:田淵ら (2002) 特願2003-45191
