効率の高いDNAマーカー選抜を可能にするSNP判別PCRマーカー
※アーカイブの成果情報は、発表されてから年数が経っており、情報が古くなっております。
同一分野の研究については、なるべく新しい情報を検索ください。
要約
SNP判別PCRマーカーは、稲の品種・系統間の一塩基のゲノムの違いをPCR法で識別できるDNAマーカーである。従来のマーカーよりも多くの多型部位にマーカーを設定でき、簡易DNA抽出法と組み合わせることで、大量のサンプルの遺伝子型を安定かつ迅速に判別することができる。
- キーワード:DNAマーカー、SNP、PCR法、簡易抽出法
- 担当:中央農研・北陸地域基盤研究部・稲遺伝解析研究室
- 連絡先:電話025-526-8251、電子メールkeko1@affrc.go.jp
- 区分:関東東海北陸農業・北陸・生物工学
- 分類:科学・参考
背景・ねらい
DNAマーカー選抜育種の普及には、育種現場で導入が容易な選抜用マーカーの開発が不可欠である。近年、ゲノム塩基配列の利用の一環として、ヒトを中心に一塩基多型(SNP)を判別できるDNAマーカーが開発されつつある。SNP判別マーカーは、選抜形質の遺伝子の近傍に構築が容易で、大規模な解析に適している等の優れた特徴を持ち、育種場面での活用が期待されている。特に利便性の高いSNP判別PCRマーカーは、判別プライマーの3’端をSNP位置に設定し、そのミスマッチを利用した遺伝子型判別法であり、実践的な育種現場で適用可能な方法ではあるが、判別の信頼性に課題を残している。そこで、このマーカーに改良を加え、簡便で安価なSNP判別PCRマーカーの基盤技術を確立する。
成果の内容・特徴
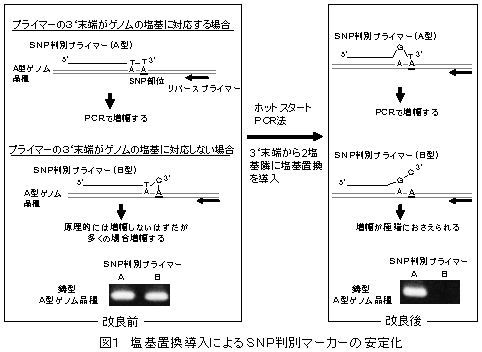
- SNP判別マーカーのプライマーの3’端のミスマッチに加え、3’端から2塩基隣に人工的に塩基置換を導入することにより、マーカーの遺伝子型判別の信頼性を上げ、さらに、プライマーのミスアニーリングを防ぐため、ホットスタートPCR法(クールスタートPCR法)を併用することにより、マーカーの安定性が向上する(図1)。
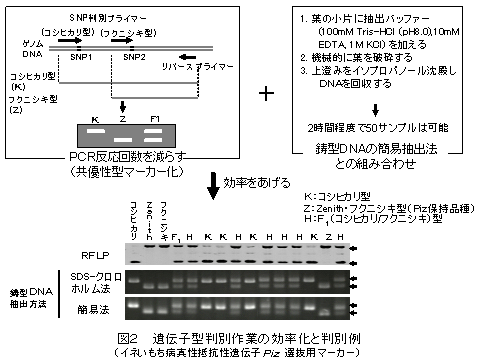
- 改良型のSNP判別PCRマーカーは、PCR産物の長さを変えた2カ所のSNP判別マーカーを混合することにより、一度のPCRでF1、F2などの交配後代系統の遺伝子型を判別することができる(図2)。
- イネの葉の小片を機械的に破砕し、イソプロパノール沈殿を行うだけの簡易DNA抽出法と改良型SNP判別PCRマーカーとを組みあわせることにより、安定で精度が高く、効率的なDNAマーカー選抜を行うことができる(図2)。
- 以上の方法を用いて、イネいもち病真性抵抗性遺伝子PizとPiz-t遺伝子のごく近傍にマーカーを設定し、マーカー選抜における有用性を検証し、確認している(図2)。
成果の活用面・留意点
- プライマーのTm値は60度以上にする。また、2カ所のSNP判別マーカーを組み合わせる場合は、PCR産物の長さの差が100bp程度になるように設計する。
- PizとPiz-t遺伝子選抜用SNP判別PCRマーカーは、コシヒカリ以外の日本の栽培稲数品種に対しても利用可能であるが、実際に選抜に使用する際は親品種で確認する必要がある。
具体的データ
その他
- 研究課題名:イネのいもち病抵抗性に関するPCR-basedのDNAマーカー選抜技術の開発
- 予算区分:DNAマーカー
- 研究期間:2001~2002年度
- 研究担当者:林敬子、橋本憲明(新潟農総研)、大源正明(新潟農総研)、吉田均、田淵宏朗、芦川育夫
