高密度遺伝子連鎖地図を高速に構築できるソフトウェアAntMap
※アーカイブの成果情報は、発表されてから年数が経っており、情報が古くなっております。
同一分野の研究については、なるべく新しい情報を検索ください。
要約
AntMapは、アントコロニー最適化を応用した新開発アルゴリズムによって高速に遺伝子座順序付けを行うことができる。これにより、高速・高精度に高密度遺伝子連鎖地図を構築することが可能となる。
- キーワード:アントコロニー最適化(Ant Colony Optimization, ACO)、遺伝子座順序付け
- 担当:中央農研・データマイニング研究チーム
- 連絡先:電話029-838-7025、電子メールiwatah@affrc.go.jp
- 区分:共通基盤・情報研究
- 分類:研究・普及
背景・ねらい
近年の分子マーカー分析技術の向上により、様々な作物で大量マーカーを利用した高密度連鎖地図作成が可能となっている。マーカー数が多い場合、マーカー遺伝子座の可能な順序の数は天文学的な数となり、現実的な時間で最適な順序を求めることが難しい。多数のマーカーを含んだ高密度連鎖地図を作成するためには、高効率アルゴリズムの開発とそのアルゴリズムを実装したソフトウェアが不可欠となる。そこで、組合せ最適化問題の解法アルゴリズムとして有効性が示されているアントコロニー最適化(Ant Colony Optimization, ACO)を応用して、遺伝子座順序付けのための新しいアルゴリズムを開発した。また、新開発アルゴリズムを実装することにより、高速に高密度遺伝子連鎖地図を構築できるソフトウェアAntMapを開発した。
成果の内容・特徴
- 新たに開発されたアルゴリズムは、多数の遺伝子座の並び順を高精度に推定可能である(表1)。
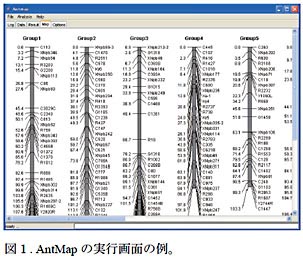
- AntMapは、グラフィカルユーザーインターフェース(GUI)を備えた、様々なOS(Windows, Linux, Solaris, Mac OSなど)上で実行可能なソフトウェアである(図1)。
- AntMapでは、連鎖地図作成に関する一連の作業:「分離比検定」、「組換え価推定」、「遺伝子座の連鎖群へのグルーピング」、「遺伝子座順序付け」、「連鎖地図の描画」が実行できる。なお、これら一連の作業は完全に自動化されており、「Full Course」メニューを選ぶと一連の作業を一挙に行うことができる。
- 遺伝子座の順序付けでは、隣接する遺伝子座間の組換え価の総和(SARF)の最小化という基準と、対数尤度(LL)の最大化という基準のどちらかを選択できる。
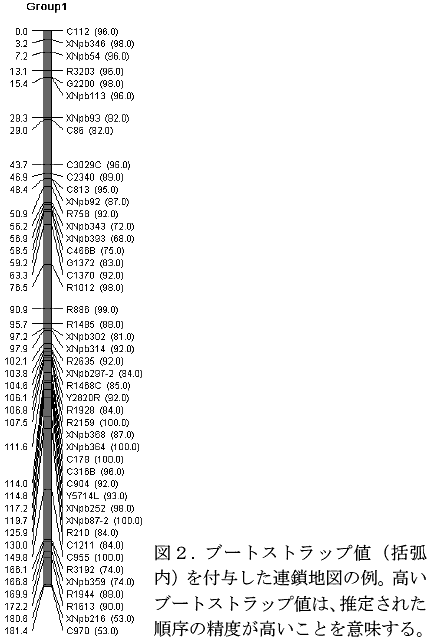
- AntMapでは、高速アルゴリズムを採用している利点を生かし、作成された連鎖地図の精度をブートストラップ検定によって確認できる(図2)。
- AntMapでは高速に連鎖地図を作成できる。例えば、6染色体に112遺伝子座が座乗した連鎖地図は、およそ22秒で構築される(InTel Pentium M 1.6GHz)。
成果の活用面・留意点
- 分離世代のマーカー遺伝子座データをMAPMAKER形式で入力する。ただし、F3(F3 self)ときょうだい交配による組換え近交系(RI-sib)には対応していない。
- 実行にはJavaプラットフォーム(J2SE JRE)が必要である。
具体的データ

その他
- 研究課題名:多様かつ不斉一なデータの融合によるデータマイニング技術の開発
- 課題ID:222-c
- 予算区分:科研費、基盤
- 研究期間:2004~2006年度
- 研究担当者:岩田洋佳、二宮正士
- 発表論文等:(1) Iwata H, Ninomiya S (2006) AntMap: constructing genetic linkage maps using an ant colony
optimization algorithm. Breed Sci 56: 371-378
