イネいもち病真性抵抗性遺伝子型を効率的に識別するDNAマーカーセット
※アーカイブの成果情報は、発表されてから年数が経っており、情報が古くなっております。
同一分野の研究については、なるべく新しい情報を検索ください。
要約
2組のマルチプレックスマーカーにより、8種類の判別品種由来のイネいもち病真性抵抗性遺伝子(Pita-Pita-2、Pik-m、Piz-t、Pik-p、Pit、Piz、Pib 、Pik)が導入された品種や同質遺伝子系統の持つ真性抵抗性遺伝子の種類を簡便に識別できる。
- キーワード:イネいもち病真性抵抗性遺伝子、DNAマーカー、マルチプレックスPCR
- 担当:中央農研・病害抵抗性研究チーム
- 連絡先:電話025-526-8251、電子メールnarc-seika@naro.affrc.go.jp
- 区分:作物、関東東海北陸農業・生物工学
- 分類:研究・普及
背景・ねらい
近年、イネいもち病真性抵抗性遺伝子型の異なる数種の同質遺伝子系統を混合栽培するマルチラインが注目を集めており、判別品種由来のイネいもち病真性抵抗性遺伝子を導入した品種、系統の育成が進められている。その一方、いもち病菌接種による検定では、複数種類の真性抵抗性遺伝子が導入された品種、系統を厳密に維持、管理するのは困難であるため、育成した品種や同質遺伝子系統を簡便に識別できるDNAマーカーが求められている。そこで本研究では、簡便に真性抵抗性遺伝子型を識別するマルチプレックスマーカーを開発する。
成果の内容・特徴
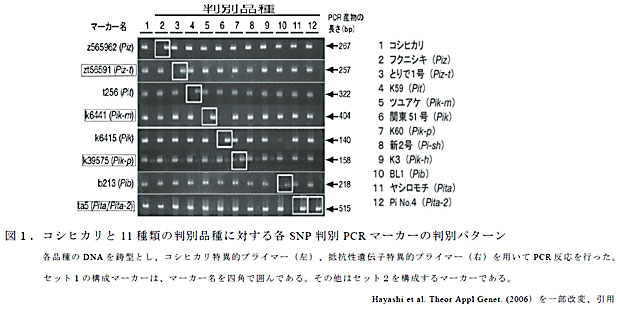
- 本DNAマーカーセットは、8種類のいもち病真性抵抗性遺伝子近傍に構築したSNP(一塩基多型)判別PCRマーカーを再構築、混合し、検出の効率化を図ったものである(表1、図1)。
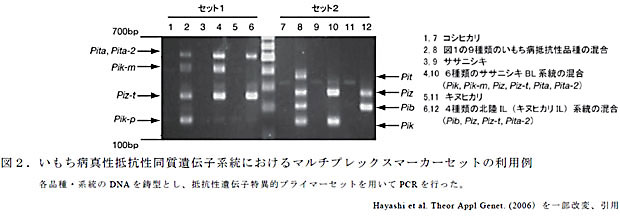
- ta5(Pita/Pita-2)、k6441(Pik-m)、zt56591(Piz-t)、k39575(Pik-p)を混合したセット1、及び、t256(Pit)、z565962(Piz)、b213(Pib) 、k6415(Pik)を混合したセット2よりなる検出マーカーセットを使用することにより、2回のPCR反応で9種類のいもち病真性抵抗性遺伝子型を識別することができる(図2)。
- 本マーカーセットを用い、5種類のササニシキBL系統(BL1(Pik)、BL2(Pik-m)、BL3(Piz)、BL4(Piz-t)、BL5(Pita-2)/BL6(Pita))、及び、4種類の北陸IL系統(キヌヒカリ;IL1(Pita-2)、IL2(Piz-t)、IL3(Pib)、IL4(Piz))を識別できる(図2)。従って、数種類の真性抵抗性遺伝子を集積させた系統を育成する過程においても利用可能である。
成果の活用面・留意点
- 本マーカーは、「コシヒカリ」、「ササニシキ」、「キヌヒカリ」以外に、「ひとめぼれ」、「あきたこまち」、「ヒノヒカリ」を同質遺伝子系統親に用いた場合にも利用できる。これ以外の品種に使用する際は、親品種でバンドの増幅パターンを確認する必要がある。
- Pita(ヤシロモチ由来)とPita-2(Pi No.4由来)とは本マーカーセットによって識別することはできない。
- 本マーカーの利用に当たっては、Hayashi et al. (2006) を引用すること。
具体的データ

その他
- 研究課題名:食用稲における病害抵抗性の強化のための遺伝子単離と機作の解明
- 課題ID:221-f
- 予算区分:基盤研究費
- 研究期間:2004~2006年度
- 研究担当者:林敬子、吉田均、芦川育夫
- 発表論文等:Hayashi et al. (2006) Theor Appl Genet. 113(2):251-60
