作物遺伝資源を用いてベイジアンアソシエーション解析によりQTLを検出する方法
※アーカイブの成果情報は、発表されてから年数が経っており、情報が古くなっております。
同一分野の研究については、なるべく新しい情報を検索ください。
要約
在来種などの作物遺伝資源を用いて、その形質値とDNAマーカー多型間の関連から、交配実験を行わずに関与QTLを検出する方法。ベイズモデルにもとづき、全QTLの効果を同時に推定することにより、誤って検出されるQTLの数を大幅に抑制できる。
- キーワード:アソシエーション解析、遺伝資源、量的形質遺伝子座(QTL)、ベイズモデル
- 担当:中央農研・データマイニング研究チーム
- 代表連絡先:電話029-838-7176
- 区分:共通基盤・情報研究
- 分類:研究・普及
背景・ねらい
古くから世界各地で独自に栽培されてきた作物在来種には、病害抵抗性など、近代品種にはない有用な形質が見いだされることがある。これまでは、このような形質に関与する遺伝子の検出やその効果の推定のためには交配実験が用いられてきた。しかし、この方法では、対象とする形質の数が多くなると、時間と労力のかかる交配実験を多数行わなければならなかった。そこで、作物遺伝資源を直接材料として用いて、交配実験を行わずに、有用形質に関与するQTLの検出や、その遺伝的効果を推定できる手法を開発する。
成果の内容・特徴
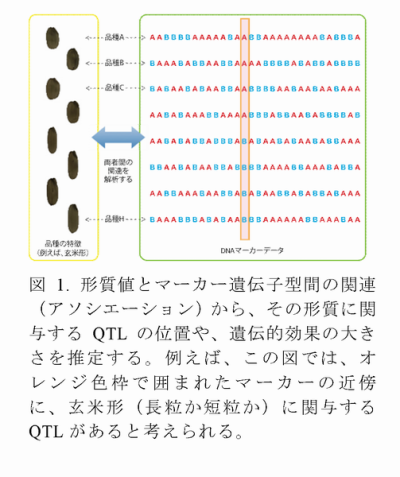
- 品種・系統の形質値と、その品種・系統から得られたDNAマーカーの多型間にみられる関連(アソシエーション)を統計的に解析することにより、その形質に関与するQTLの位置や、そのQTLが形質に及ぼす効果の大きさを推定する(図1)。
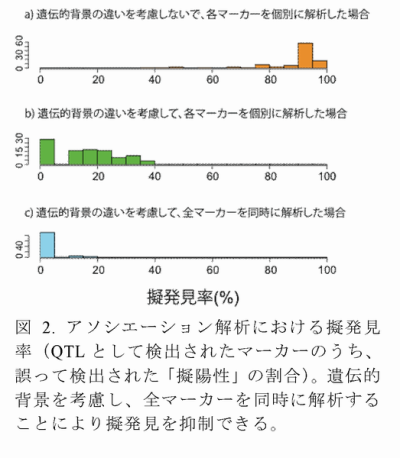
- QTLの誤検出(擬陽性)は、遺伝構造の影響により擬陽性が生じやすい作物のアソシエーション解析における最大の問題である。品種・系統間の遺伝的背景の違いを考慮し、かつ全マーカーを同時に解析する(全QTLの効果を同時に推定する)統計モデルにより、誤ってQTLとして検出されるマーカーの数を大幅に抑制できる(図2)。
- アソシエーション解析にベイズモデルを採用したことにより、統計モデルを実験の条件やデザインに合わせて柔軟に拡張できる。例えば、形質の計測値が、通常の連続データの場合だけではなく、多段階(3段階、5段階など)で評価されたデータや、限られた実験期間のために観察が途中で打ち切られたデータ(例えば、出穂期のデータで、一部の晩生品種が実験期間中に出穂しなかった場合)の場合でも、精度良くQTLを検出し、その効果を推定することができる。
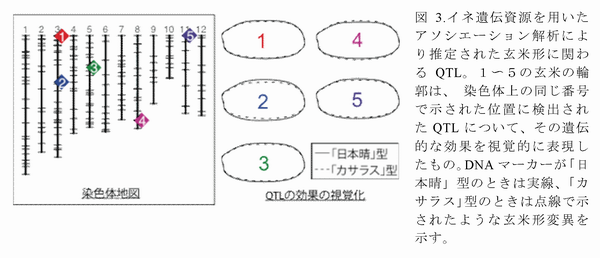
- 同手法を、“「かたち」を定量的に計測・評価するためのプログラムパッケージSHAPE” (平成15年度成果)と組み合わせて利用することにより、作物の形を支配するQTLの検出、およびその遺伝的効果の推定が可能となる(図3)。
- 同手法は、ゲノムワイドマーカーに基づき形質値を予測して選抜する方法(ゲノミックセレクション)にも利用できる。
成果の活用面・留意点
- 形質データ、マーカーデータ、遺伝的背景データをもとにアソシエーション解析を行うJavaプログラムを作成した。同プログラムは、現在、共同研究を条件に利用できる。将来はフリーソフトとして配布の予定である。
- DNAマーカーとして、一塩基多型(SNP)のような二対立遺伝子のものと、マイクロサテライト(SSR)のように複対立遺伝子のものの両方を扱うことができる。
- 栽培イネのような自殖性の植物だけでなく、果樹や林木などのように他殖性で、マーカーやQTLがヘテロ接合になっている植物にも適用できる。
具体的データ
その他
- 研究課題名:多様かつ不斉一なデータの融合によるデータマイニング技術の開発
- 中課題整理番号:222c
- 予算区分:基盤、委託プロ(QTL)、委託プロ(他作物マーカー)、科研費
- 研究期間:2006~2009年度
- 研究担当者:岩田洋佳、江花薫子(生物研)、林武司(生物研)
- 発表論文等:1) Iwata H. et al. (2007) Theor. Appl. Genet. 114(8): 1437-1449 2) Iwata H. et al. (2009) Theor. Appl. Genet. 118(5): 865-880 3) Iwata H. et al. (2010) Mol. Breed. 25(2): 203-215
