フザリウム群集のPCR-DGGEを利用したアスパラガス連作圃場の診断指標
要約
アスパラガスに病原性を有するフザリウムを識別・解析可能なPCR-DGGE法で検出されるバンド全体に占めるFusarium. oxysporum f.sp. asparagi由来のバンド強度の割合は連作障害発生圃場で高い。そのバンド強度の割合が高い圃場では欠株率も高い傾向にあり、圃場診断の指標となり得る。
- キーワード:アスパラガス、PCR-DGGE、フザリウム、連作障害、群集構造
- 担当:総合的土壌管理・土壌生物機能評価
- 代表連絡先:電話 029-838-8481
- 研究所名:中央農業総合研究センター・土壌肥料研究領域
- 分類:研究成果情報
背景・ねらい
アスパラガス栽培において、株を更新する場合に、経営面積の小さい我が国では同一圃場に定植(改植)を余儀なくされている。アスパラガスは改植時に欠株などの生育不良が発生するなどの問題があるが、改植前に診断する方法が無く、土壌生物性解析に基づく診断法の開発が求められている。そこで連作障害発生圃場の細菌および糸状菌群集構造とフザリウム群集構造をPCR-DGGE法を用いて解析し、圃場診断法の開発につながるアスパラガス連作障害土壌の微生物指標を明らかにすることを目的とした。
成果の内容・特徴
- 調査した圃場の土壌化学性は、全般的に可給態リン酸の値と交換性カリウムの含量が高い農家が多く、交換性カルシウムの含量も高い。しかしながら連作障害発生圃場に特徴的な化学性の要因は認められない。さらに細菌および糸状菌群集構造に関しても連作障害発生の有無で顕著な差異は認められない(データ略)。
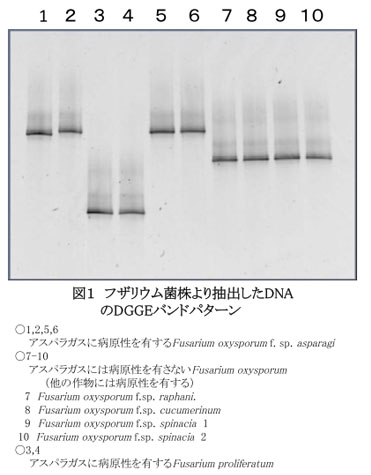
- PCR-DGGE法によるフザリウムの検出ではペプチド鎖伸長因子EF-1αを対象とし、プライマーEF1とEF2およびフザリウム特異的なプライマーAlfie1とAlfie2GCを用いてnested PCRを行う。次にPCR産物を変性剤濃度勾配(40%→60%)アクリルアミドゲルを用いて60°C、110Vで17時間電気泳動する。この条件で既知菌株より抽出したDNAを用いてPCR-DGGE解析を行うことにより、アスパラガスの病原菌であるFusarium oxysporum f.sp. asparagiと他の作物の病原菌であるF. oxysporumとを区別して解析することが可能である(図1)。
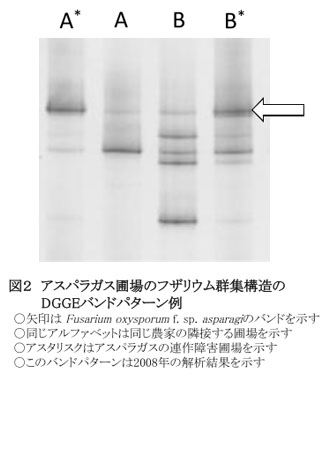
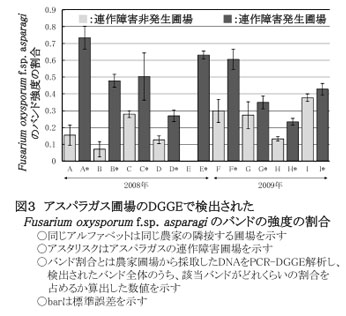
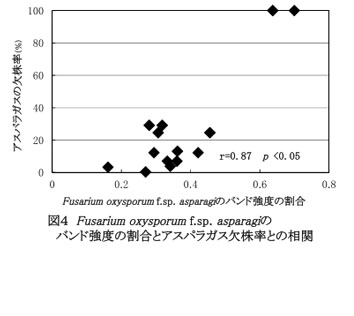
- フザリウム群集を対象としたPCR-DGGE法で検出されるバンド全体に占めるアスパラガスの病原菌であるF. oxysporum f.sp. asparagiのバンド強度の割合は連作障害発生圃場で高く(図2、図3)、そのバンド強度の割合と改植時におけるアスパラガスの欠株率との間に正の相関が認められる(図4)。同様にアスパラガスの病原菌であるF. proliferatumのバンド強度の割合については一定の傾向が認められない。そのためにF. oxysporum f.sp. asparagiのバンド強度の割合が高い圃場は改植時において欠株などの障害発生の危険度が高い傾向にある。
成果の活用面・留意点
- 本研究成果は、福島県会津盆地内全域を代表とするアスパラガスの連作障害圃場と隣接する健全生育圃場より採取した土壌を対象とした結果である。
- 圃場内のばらつきは大きいため、対角線採土法などを用いて5ヶ所以上で土壌を採取することが望ましい。また、アスパラガスの根が比較的多く存在する部位(深さ15cm-25cm)より土壌を採取する必要がある。
- PCR-DGGE法は絶対量を評価する方法ではないので、バンド強度の割合と菌密度の関係については今後検討が必要である。
- 本方法を現地に普及させる際には、さらなるデータ蓄積により、F. oxysporum f.sp. asparagiのバンド強度の割合と許容欠株率との間において指標値の関係を示す必要がある。
具体的データ
(浦嶋泰文)
その他
- 中課題名:土壌生物機能を核とした土壌生産力評価法の開発
- 中課題番号:151c0
- 予算区分:実用技術開発事業、交付金、所内活性化経費(東北研)
- 研究期間:2008~2011年度
- 研究担当者:浦嶋泰文、藤田祐子、園田高広、浦上敦子
- 発表論文等:Urashima Y. et al. (2012) Microbes and Environments 27(1):43-48
