選抜したSSRマーカーは国内分離いもち病菌の集団解析に有用である
要約
選抜した12種類のSSRマーカーを用いれば、国内分離いもち病菌を高精度に個体識別でき、検出される遺伝子型に基づきいもち病菌集団の遺伝的多様度や集団間の遺伝的類縁関係を推定できる。
- キーワード:SSRマーカー、イネいもち病、集団遺伝学的解析、マルチプレックス化
- 担当:環境保全型防除・水稲病害抵抗性
- 代表連絡先:電話 029-838-8481
- 研究所名:中央農業総合研究センター・病害虫研究領域
- 分類:研究成果情報
背景・ねらい
SSR(Simple Sequence Repeat)は、ゲノム中に広く散在する短いモチーフ(1~6塩基)の繰り返し配列であり、多型性に富み遺伝的に比較的安定、中立で共優性を示す特徴がある。このため、SSRマーカーを利用すれば、いもち病菌を個体レベルで識別してその挙動を追跡する、あるいはいもち病菌を集団として捉えてその変動を集団遺伝学的に解析することが可能となる。本研究では、いもち病菌のゲノム情報に基づきSSRマーカーの候補領域を探索し、国内分離菌の解析に有効なマーカーを選抜する。選抜したSSRマーカーについては、2001年西日本分離菌を対象として、マーカーごとのアリル数や集団の遺伝的多様度などの基本的なパラメータを算出する。
成果の内容・特徴
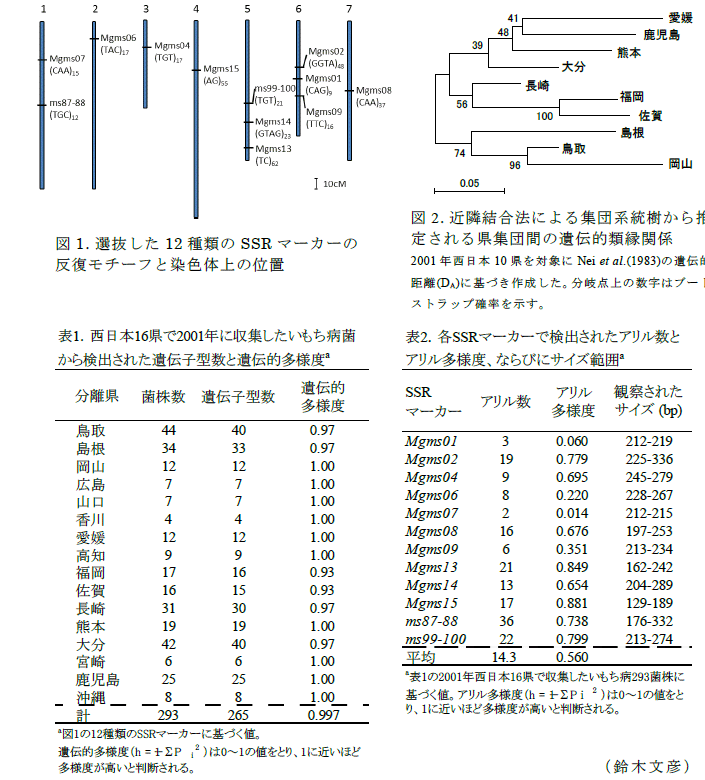
- SSRマーカーの探索は、Magnaporthe grisea Database の塩基配列情報を活用し、反復配列探索ソフトTandem Repeats Finder (Benson 1999)で実施する。多型性や座乗位置などを考慮して選抜した12種類SSRマーカーの染色体上の位置を図1に示す。
- 2001年に西日本16県から収集したいもち病293菌株を対象に、選抜した12種類のSSRマーカーを組み合わせて解析した結果、265の遺伝子型に類別され遺伝的多様度は0.997となり、高精度な個体識別が可能である(表1)。また、マーカーごとの検出アリル数は2~36、アリル多様度は0.014~0.881と大きく異なり、SSR座によって変異頻度に違いのあることが推定される(表2)。
- 解析菌株数が10以上の10県を対象にして近隣結合法で集団系統樹を作成すると、中国地方の3県、九州の3県、四国と九州の4県がそれぞれクラスターを形成し、近距離の県集団間で遺伝的距離が小さい傾向を示す(図2)。
- 本マーカーは、同一品種間および近接圃場間のいもち病菌集団の識別にも有効であることを確認している(データ未掲載)。
成果の活用面・留意点
- 秋田県立大学バイオテクノロジーセンターで12種類のSSRマーカーの解析を受託している。解析費用は96サンプルで7万円程度である。
- キャピラリーシーケンサーを用いて解析する場合、PCR反応をマルチプレックス化(各4マーカーを含む3セット)すれば、作業効率を大幅に向上できる。
- SSR座によって変異頻度に差があることが推定され、研究目的に応じて使用するマーカーの組み合わせを選択する必要がある。
- 本マーカーを集団遺伝学的パラメータ(選択圧、移出入率、遺伝的浮動など)の解析に使用することを予定している。
具体的データ
その他
- 中課題名:水稲病害抵抗性の持続的利用技術の開発
- 中課題番号:152c0
- 予算区分:交付金
- 研究期間:2009~2012
- 研究担当者:鈴木文彦、善林 薫、藤 晋一(秋田県大)、荒井治喜、中島 隆
- 発表論文等:
1)Suzuki, F. et al. (2009) Mol. Ecol. Resour. 9: 588-590
2)鈴木ら (2012) 日植病報、78: 10-17
