豚レンサ球菌強毒株菌体表層に形成されるSrtG線毛の証明とその組立て機構
要約
豚レンサ球菌強毒株を識別する遺伝子マーカーの1つであるsrtG遺伝子群を持つ菌株は表層に線毛(SrtG線毛)を形成する。SrtG線毛は組立てに線毛修飾蛋白質を必要とする新しいグラム陽性菌の線毛である。
- キーワード:豚レンサ球菌、グラム陽性菌、線毛、線毛の組立て
- 担当:動物衛生研・細菌・寄生虫病研究チーム
- 代表連絡先:電話029-838-7708
- 区分:動物衛生
- 分類:研究・参考
背景・ねらい
線毛は細菌の菌体表層に分布する毛様の蛋白質複合体で、多くの病原細菌において感染の成立に重要な宿主への定着に寄与する。豚レンサ球菌症の病原体である豚レンサ球菌(Streptococcus suis)の強毒株にも線毛の形成に関与する関連遺伝子群がsrtG領域に見出され、毒性の識別に利用されているが、その発現産物である線毛は未だに見出されていない。本研究ではsrtG領域について、線毛形成及びその組立て機構の詳細を調べる。
成果の内容・特徴
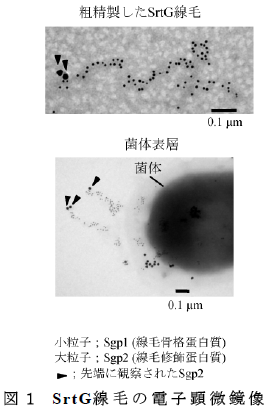
- S. suisのsrtG領域は線毛骨格蛋白質(Sgp1)、線毛修飾蛋白質(Sgp2)及びそれらの構築に関与する組立て酵素(SrtG)をコードする3つの遺伝子で構成される。標識抗体を用いた電子顕微鏡観察により、強毒株であるS. suis 89/1591株の菌体表層に、骨格がSgp1から成り、先端にSgp2が付加した線毛様構造が観察できる(図1)。
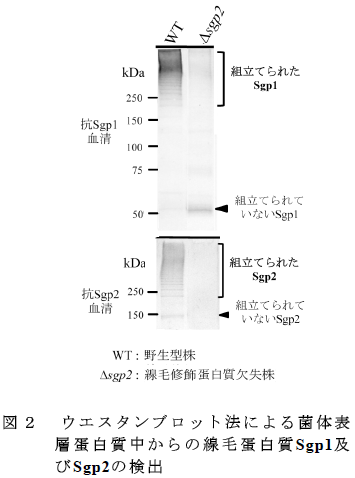
- 特異抗体を用いたウエスタンブロット法により菌体表層の線毛蛋白質を検出すると、様々な構成比に応じた高分子量のラダーとして観察される。他のグラム陽性菌種では、線毛修飾蛋白質がなくても線毛が形成されるが、SrtG線毛の組立てには線毛修飾蛋白質が必要であり、線毛修飾蛋白質欠失株では高分子量ラダーが消失する(図2)。
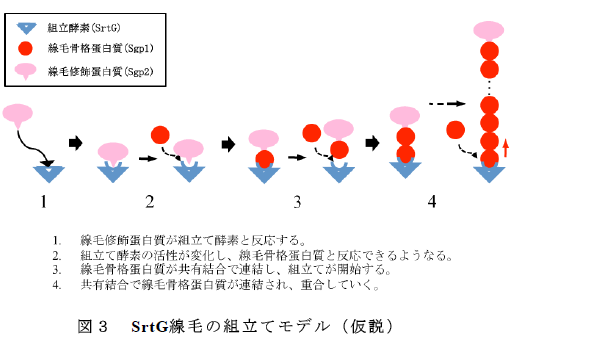
- グラム陽性菌の線毛は線毛蛋白質と組立て酵素の間に別の線毛蛋白質が付加し、連結していく事で構築される。SrtG線毛では、Sgp2は先端に位置する事から組立てを開始する引き金となる分子であると考えられる(図3)。
- 複数のグラム陽性菌種でもSgp2に相同性を有する蛋白質が存在する事から、一部のグラム陽性菌の線毛では、Sgp2と同様に線毛修飾蛋白質が線毛形成に寄与する可能性を示している。
成果の活用面・留意点
- 本研究成果により、S. suis強毒株の菌体表層におけるSrtG線毛の発現が証明され、強毒株の識別用マーカーとして簡易診断法への応用が期待できる。
- 他の病原細菌の線毛と同様にSrtG線毛が宿主への定着に寄与するかについてはさらに解析を進める必要がある。
- 本研究成果で提案した新たな線毛の組立てモデルを証明するためには、線毛蛋白質間ならびに組立て酵素との相互作用の詳細を明らかにする必要がある。
具体的データ
その他
- 研究課題名:細菌・寄生虫感染症の診断・防除技術の高度化
- 中課題整理番号:322e
- 予算区分:科研費
- 研究期間:2008~2009年度
- 研究担当者:大倉正稔、大﨑慎人、関崎 勉(東京大農)、Nahuel Fittipaldi(モントリオール大獣医)、Marcelo Gottschalk(モントリオール大獣医)、髙松大輔
- 発表論文等:Okura, M. et al. (2011) J. Bacteriol. 193 (4): 822-831
