牛由来Salmonella Typhimuriumの遺伝子型に基づくデータベースの構築
要約
Multiple-locus variable-number tandem-repeat analysis (MLVA)およびパルスフィールドゲル電気泳動 (PFGE)プロファイルに基づくデータベースを用いることにより、流行型のモニタリングや新型菌の検出が可能となる。MLVAによる識別能力はPFGEと比較して高いが、両者を併用することで、菌株間の関連性をより詳細に解析することが可能となる。
- キーワード:Salmonella Typhimurium、Salmonella O4:i:-、PFGE、MLVA
- 担当:家畜疾病防除・大規模酪農衛生
- 代表連絡先:電話 029-838-7708(情報広報課)
- 研究所名:動物衛生研究所・寒地酪農衛生研究領域
- 分類:研究成果情報
背景・ねらい
Salmonella Typhimurium (ST) は、子牛だけでなく搾乳牛にもサルモネラ症を引き起こし、酪農経営上大きな問題となる。流行株の特徴や感染源・感染経路等を迅速に把握することが、防疫対策を講じる上で重要であり、そのための技術・手法の確立が必要となる。本研究では、菌株間の関連性を解析する手法として、ゲノムDNAを制限酵素で切断し、そのプロファイルを比較するパルスフィールドゲル電気泳動 (PFGE)と併せて、データベース化が容易な方法として、ゲノム中の縦列反復配列数を解析するmultiple-locus variable-number tandem-repeat analysis (MLVA) 法を用い、牛由来STおよびSalmonella O4:i:- の型別を実施する。
成果の内容・特徴
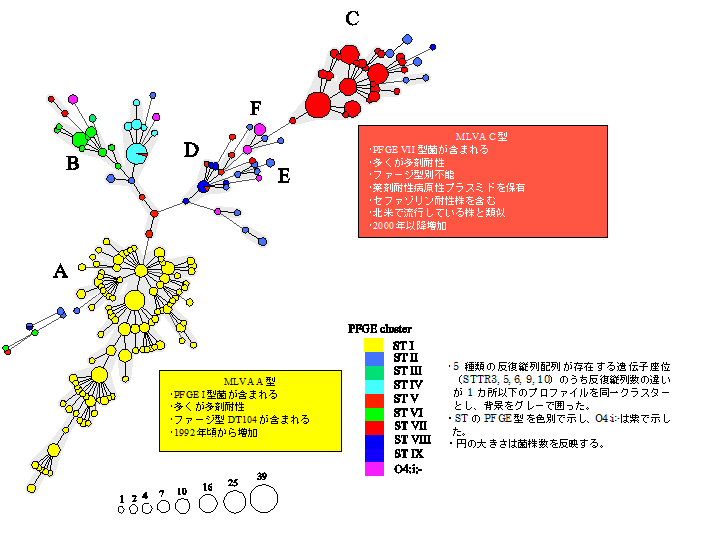
- MLVAによりST 544株およびO4:i:- 18株から184種類のプロファイルが検出され、これらは6種類の主要なクラスター (A~F) に分類される(図)。
- クラスターAの菌株は、成牛のサルモネラ症の増加に伴い1992年ごろから分離数が増加した多剤耐性ファージ型104 (DT104) を含むPFGE I型の菌とほぼ一致した。クラスターCには2000年以降新たに出現し、薬剤耐性遺伝子が乗った病原性プラスミドを保持する多剤耐性PFGE VII型菌が含まれる。
- O4:i:- からは、5種類のPFGEプロファイルと6種類のMLVAプロファイルが検出される。STと同一のプロファイルを示すものは認められないが、クラスターFには極めて近いプロファイルを示すST株がある(図)。
- MLVAによる識別能力(DI値0.982)はPFGE(DI値0.852)よりも高くなるが、PFGE型ごとに比較すると、MLVAよりもPFGEによる識別能力が高くなる場合がある。
- MLVAとPFGEを併用すると、それぞれを単独で解析した場合と比較して識別能力が高くなる(表)。
成果の活用面・留意点
- 1990年以降、多剤耐性を示す2種類のMLVA 型(AおよびC型)が新たに出現し、現在分離される殆どのST株がこれらに含まれ、耐性株の広がりに注意を要する。
- MLVAとPFGEを併用することにより、STおよびO4:i:- における菌株間の関連性をより詳細に解析することが可能となり、感染源・感染経路の特定に利用することができる。
- MLVA、PFGE、および薬剤耐性のプロファイルを用いたデータベースの構築により、流行型および薬剤耐性のモニタリング、新型菌出現の検出等が可能となる。
- 当面は本データベースを動衛研において、病性鑑定に活用する。将来的には、インターネット上での情報共有システムの構築を検討する。
具体的データ
その他
- 中課題名:乳房炎等の大規模酪農関連疾病の診断・防除法の開発
- 中課題番号:170e1
- 予算区分:委託プロ(生産工程)
- 研究期間:2008~2012年度
- 研究担当者:玉村雪乃、内田郁夫、田中聖、秋庭正人、秦英司、菅野徹、黒澤篤(北海道)、今村毅 (北海道)、小林亜由美(北海道)、湯川尚一郎(酪農学園大)、田村豊(酪農学園大)
- 発表論文等:Kurosawa A. et al. (2012) Vet. Microbiol. 160(1-2):264-268
