豚丹毒菌生ワクチン株と野生株とを識別できるPCR法の開発
要約
豚丹毒菌生ワクチンKoganei 65-0.15株ゲノム塩基情報のわずかな違いをPCR法により検出することで、本株を特異的かつ迅速に同定することができる。
- キーワード:豚丹毒菌、生ワクチン株、ゲノムSNPs、同定
- 担当:家畜疾病防除・先端的疾病防除技術
- 代表連絡先:電話 029-838-7708
- 研究所名:動物衛生研究所・細菌・寄生虫研究領域
- 分類:普及成果情報
背景・ねらい
豚丹毒はグラム陽性細菌の豚丹毒菌(Erysipelothrix rhusiopathiae)による豚の感染症で、家畜伝染病予防法において届出伝染病に指定されている。本病は、症状から、急性の敗血症型、亜急性の蕁麻疹型、慢性の関節炎型や心内膜炎型に大別される。国内では、毎年2,000?3,000頭の発生報告があり、養豚産業に及ぼす経済的影響は甚大である。豚丹毒の予防には、生ワクチン及び不活化ワクチンが使用されているが、生ワクチン使用による慢性関節炎の発生が問題となっている。現在、国内で使用される生ワクチンはアクリフラビン色素耐性の弱毒株である Koganei 65-0.15株(血清型1a)のみであり、形質マーカーとされるアクリフラビン色素耐性は継代により変化することから、関節炎病変部から分離される野外株の血清型が1aであった場合、この株が野生株であるか、本ワクチン株であるかどうかの識別は困難である。生ワクチン株を正確に識別できれば、ワクチンプログラムの変更を促す等の技術指導が可能になり、生ワクチン使用による慢性関節炎の発生を予防することが期待できる。本研究では、Koganei 65-0.15株を特異的かつ迅速に同定することができる技術を開発する。
成果の内容・特徴
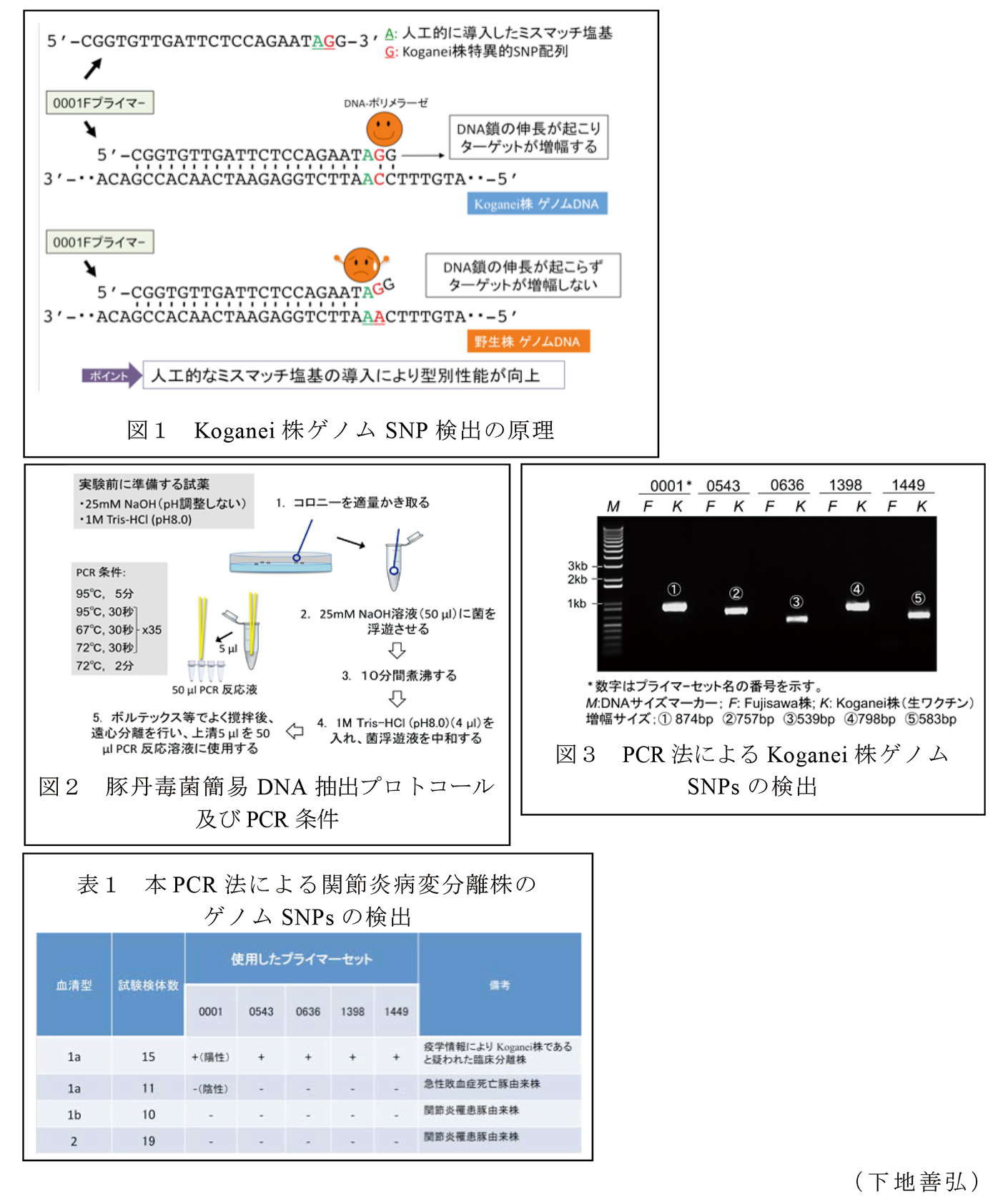
- 設計した5組のプライマーセットは、Koganei 65-0.15株(血清型1a、以下 Koganei株)のゲノムドラフト配列とデータベースに登録されているFujisawa 株(血清型1a)の全ゲノム配列との比較解析により見出した一塩基多型(SNPs)のうち、ランダムに選んだ5個をターゲットとし、Koganei株に特異的なSNPsでのみ遺伝子断片の増幅が起こるようにしたものである。
- これらのプライマーでは、擬陽性の発生を抑制するため、Forward プライマーの3'側から2番目と3番目の塩基に、それぞれ検出したいSNPと人工的に導入したミスマッチの塩基を置くように設計されている(図1)。
- PCR反応に用いる鋳型DNAには、菌をアルカリ溶液で煮沸し、中和した後に遠心分離した上清を用いる(図2)。
- 設計した5組のプライマーセットを用いてPCRを行うと、Koganei 株とFujisawa 株を識別可能である(図3)。また、関節炎症状を示す豚から分離された、疫学情報からKoganei株であると疑われる臨床株15株についてはすべて増幅する(表1)。
普及のための参考情報
- 普及対象:家畜保健衛生所等の病性鑑定担当者、食肉検査所等の検査担当者
- 普及予定地域・普及予定面積・普及台数等:全国
- その他:本検出法は、プルーフリーディング(3→5′エクソヌクレアーゼ)活性及び遺伝子増幅能が高いKOD-FX等の酵素を用いることが重要である。
- 使用するサーマルサイクラーによっては、一部のバンドが増幅されないことがあるので、アニーリング温度を上下に変更調節し、最適温度を決定する。
具体的データ
その他
- 中課題名:先端技術を利用した新しい疾病防除技術の確立
- 中課題整理番号:170c2
- 予算区分:交付金、委託プロ(生産システム)
- 研究期間:2012~2015年度
- 研究担当者:下地善弘、白岩和真、小川洋介、江口正浩、楠本正博
- Shiraiwa K. et al.(2015) J. Microbiol. Methods. 117:11-13
