近年我が国で流行した豚流行性下痢(PED)ウイルス株の全ゲノム配列
要約
我が国で大流行したPEDウイルス38株の全ゲノム配列は、大きく3つのグループに分類され、いずれも2013年以降の米国、メキシコなどの北米流行株と極めて近縁であることから、これらウイルスは海外から複数回にわたって侵入した可能性が考えられる。
- キーワード:PEDウイルス、次世代シークエンサー、パンデミック、全ゲノム、遺伝子欠損
- 担当:家畜重要疾病・ウイルス感染症
- 代表連絡先:電話 029-838-7708
- 研究所名:動物衛生研究所・ウイルス・疫学研究領域
- 分類:研究成果情報
背景・ねらい
2013年10月以降、我が国では7年ぶりに豚流行性下痢(PED)が流行し、これまでに約42万頭の豚が死亡した。PEDは、2013年以降、米国、カナダ、メキシコの北米各国、中国、韓国、ベトナムなどのアジア各国、さらにドイツ、フランス、ベルギーなどの欧州各国でも発生し、パンデミックの様相となっている。本研究では、次世代シークエンサーを活用して、近年の国内流行PEDウイルス株の全ゲノムを解読し、既知の国外流行株と全配列を比較・解析することで、国内流行株の由来を推察する。
成果の内容・特徴
- 次世代シーケンサーを用いて、PEDウイルスの全ゲノムを同時に且つ迅速に解読する方法を開発し、2013年以降本病が流行した18県から集められた国内流行PEDウイルス38株の全ゲノムを解読する。
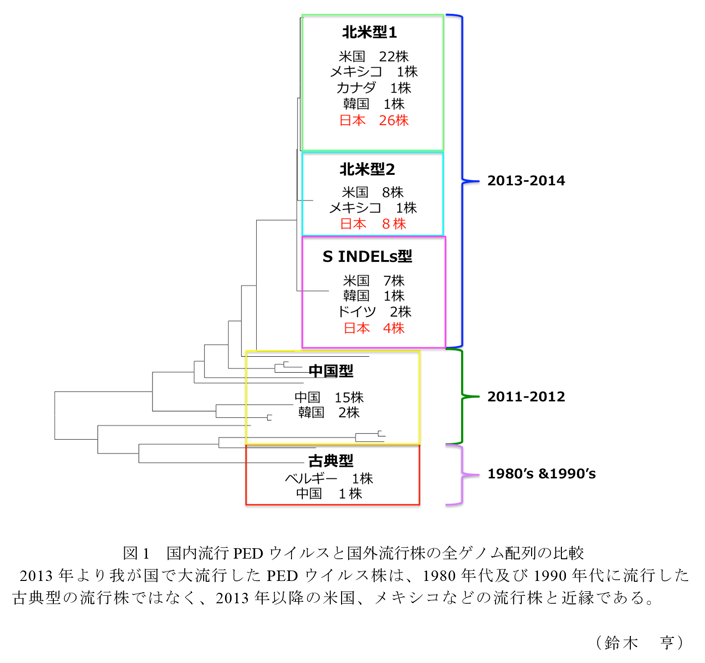
- 国内流行PEDウイルス38株と米国、メキシコ、中国及び韓国などの国外流行株120株の全ゲノムを比較・解析したところ、近年の国内流行株は2013年以降の米国、メキシコなどの北米流行株と遺伝的に極めて類似する一方、2011年から2012年にかけて中国で流行した株及び1970年代後半から1980年代にかけてベルギーや中国で流行した株とは遺伝的に明確に区別される(図1)。
- 近年の国内流行PEDウイルス株を既知の報告に従って分類した結果、26株は北米型1、8株は北米型2、残りの4株は、INDELs型(北米型と比べて2箇所の欠損と1箇所の挿入を特徴とする)の3つの遺伝的グループに分類される。この結果は、近年の国内流行PEDウイルス株の侵入経路が単一ではなく、海外から複数回にわたって侵入した可能性を示す。
成果の活用面・留意点
- 次世代シーケンサーにより、国内流行PEDウイルス株の全ゲノムを迅速に解読して、それらの由来を推察することは、我が国のPED防疫対策を構築する一助となる。
- 株間における全ゲノムを比較して、特定の株間における特異的な保存領域(分子疫学マーカー)を探索し、ウイルスの流行動態や流行予測に役立てる。
- 株間におけるゲノム配列の相違とウイルス株特有の病原性や組織局在性の相違を関連付けることで、各遺伝子の機能を推測する。
具体的データ
その他
- 中課題名:ウイルス感染症の発症機構の解明と防除技術の確立
- 中課題整理番号:170a1
- 予算区分:競争的資金(農食事業)
- 研究期間:2014年度
- 研究担当者:鈴木亨、宮﨑綾子、大橋誠一、筒井俊之、小寺文(宮城県)、増田恒幸(鳥取県)、村上聡(ライフテクノロジーズジャパン株式会社)、高橋理(ライフテクノロジーズジャパン株式会社)
- 発表論文等:Suzuki T. et al. (2015) Infection, Genetics and Evolution 36:363-368
