SSRマーカーによるシバの品種識別技術
※アーカイブの成果情報は、発表されてから年数が経っており、情報が古くなっております。
同一分野の研究については、なるべく新しい情報を検索ください。
要約
シバ品種「朝駆」のゲノムライブラリーから開発したSSRマーカーは、シバ品種・系統を効率よく識別できる。また、他の種でもバンドの増幅が見られ、シバ属に広く利用できる。
- キーワード:飼料作物育種、イネ科牧草、シバ、SSR、品種識別
- 担当:畜産草地研・飼料作物開発部・育種資源研究室
- 連絡先:電話0287-37-7552、電子メールkobamako@affrc.go.jp
- 区分:畜産草地、作物(生物工学)
- 分類:技術・普及
背景・ねらい
種苗法に基づく育成者権保護制度の制定にともない、品種の区別性や均一性を検証するための迅速・簡易な品種識別技術の早期確立が強く求められている。特 に、類似品種の判別に有効な指標形質が少なく、かつ、栄養繁殖によって容易に増殖することが可能なシバでは、信頼性の高い客観的な識別技術の開発は急務と なっている。本課題は、ゲノム中に散在する単純反復配列(SSR)を利用した再現性の高いDNAマーカーを開発し、それを利用したシバの品種識別技術の確 立を目的とする。
成果の内容・特徴
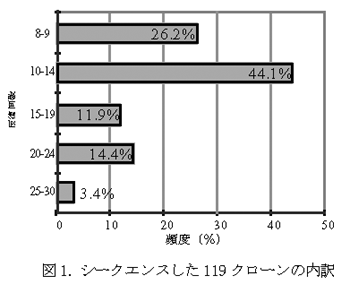
- シバ品種「朝駆」由来のAG/TC濃縮ゲノムライブラリーから160クローンを選抜し、そのうち119クローンでSSR領域が認められた。119のSSR領域における反復回数は、8-28回の範囲であった(図1)。
- 上記119種類のうち32種類のSSR領域についてプライマーを設計し、これをもとに23種類のSSRマーカーを開発できた。
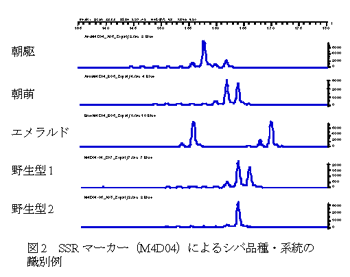
- 「朝駆」を含むシバ5品種・系統を用いてSSR解析を行った。供試した全ての品種・系統で異なるバンドパターンが得られ、効率よく各品種・系統の遺伝子型を判定することができる(図2)
- 開発したSSRマーカーは、シバのみならず、コウライシバ、コウシュンシバおよびオニシバでも増幅することが認められ、シバ属に広く適用できる(表1)。
成果の活用面・留意点
- 客観的で信頼性の高いシバ品種の識別が可能となる。
- 新たな育成品種にも迅速に対応するため、SSRマーカーによって既存品種・系統の遺伝子型を整理する必要がある。
- 1クローン由来の栄養繁殖性品種以外では、別途検討が必要である。
具体的データ

その他
- 研究課題名:暖地型牧草の重要形質に関する連鎖解析に向けたDNAマーカーの開発
- 課題ID:12-05-02-01-19-03
- 予算区分:交付金
- 研究期間:2003~2005年度
- 研究担当者:霍田真一、高原 学、蝦名真澄、中川 仁
- 発表論文等:特許出願「シバ属植物の個体識別方法」特願2003-271137
