ルーメン内容物から抽出したDNAによるルーメン細菌の同定
要約
ルーメン細菌の働きを調べるため、ルーメン内容物から採取したDNA配列を自己組織化図法(SOM法)で解析すると、ルーメン細菌として確認されていない細菌種が見つかり、ルーメンには多くの未同定細菌が存在・機能していることが明らかになる。
- キーワード:ルーメン細菌、遺伝子、自己組織化図法(SOM法)、未培養細菌
- 担当:畜産草地研・分子栄養研究チーム
- 代表連絡先:電話029-838-8611
- 区分:畜産草地
- 分類:研究・参考
背景・ねらい
ルーメン細菌叢による飼料分解(ルーメン発酵)はウシに栄養を供給するための重要な消化活動であり、より効率的なルーメン発酵をめざすにはルーメン細菌をよく知る必要がある。しかし、大多数のルーメン細菌は培養不可能な細菌であり、ルーメン細菌叢を構成する細菌を特定することは困難である。
そこで、遺伝子配列情報から細菌種を類推することができる自己組織化図法(SOM法)を用いてルーメン内容物から分離されている多くの遺伝子を解析し、ルーメン細菌叢を構成する細菌種の同定を検討する。
成果の内容・特徴
- DNAデータベース(NCBI (http://www.ncbi.nlm.nih.gov/))で公開されている細菌および古細菌のゲノム情報(602菌種分)を収集し、各ゲノム配列からランダムに1MbのDNA配列を抽出する。このDNA配列をSOM法(Abeら(2000) Gene 365, 27–34)の原図作成に用いる。
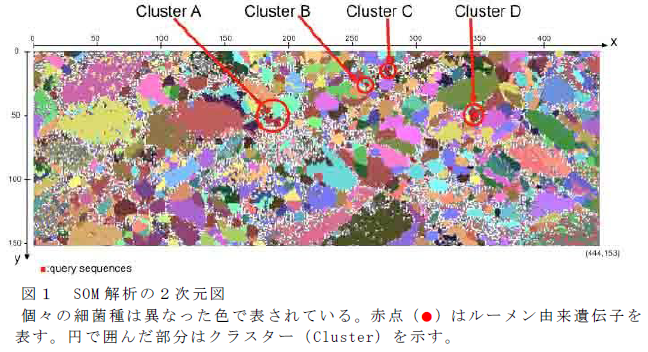
- 真核生物およびリボゾームRNA遺伝子を除いた、ルーメン由来の660のDNA配列をSOM法で解析すると、多くのDNA配列は原図上に広くプロットされるが、一部は狭い範囲に集中し、クラスター(Cluster)を形成する(図1)。
- 植物の主要な構成成分であるセルロースを分解する細菌(セルロース分解菌)として、これまでルーメン内で確認されていないClostridium phytofermentans、Clostridium thermocellum、Cytophaga hutchinsoniiが検出され、より多くの細菌種がルーメン内セルロース分解に関与していることが明らかとなる。
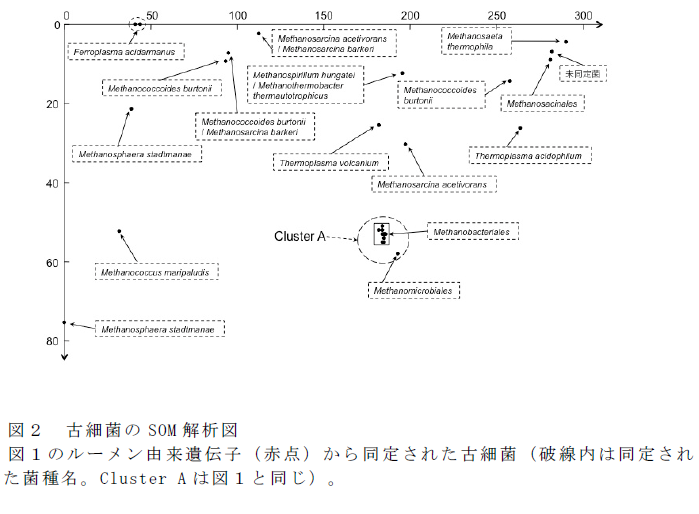
- 古細菌由来のDNA配列からは主にメタン菌が同定されたが、メタン菌ではないThermoplasmataに属する古細菌や未同定菌が見つかる(図2)。
成果の活用面・留意点
- ルーメン由来の機能性遺伝子の起源(細菌種)が同定されることから、細菌種の同定とともに、ルーメン機能とそれを担う細菌種とを関連させることができ、個々のルーメン細菌の特徴を明らかにできる。
- ルーメン細菌叢から収集される遺伝子情報は急速に増大することが予想されるが、そのほとんどが未培養細菌由来と推定される。SOM解析は遺伝子配列の起源(細菌種)を同定することができることから、ルーメン微生物相のより詳細な基礎的知見が集積される。
- SOM解析はルーメン微生物相の解析だけでなく、未培養細菌が多数存在するその他の環境微生物相への応用が可能である。
具体的データ
その他
- 研究課題名:栄養素による遺伝子発現調節機能の解明
- 中課題整理番号: 221m
- 予算区分: 基盤
- 研究期間:2006~2010年度
- 研究担当者: 三森眞琴、真貝拓三、松井宏樹(三重大)、竹中昭雄(国際農研)
- 発表論文等:Mitsumori M. et al. (2010) J. Appl. Microbiol. 109(3):763-770
