マイクロアレイデータ解析のための簡易型データベース管理システムの構築法
※アーカイブの成果情報は、発表されてから年数が経っており、情報が古くなっております。
同一分野の研究については、なるべく新しい情報を検索ください。
要約
マイクロアレイ解析で利用される多量のデータを効率的に処理し統合するため、Stand- alone BLAST、Perl及びWindowsEXCELのマクロを用いて、EXCEL をプラットホームにした簡易型データベース構築法を考案した。
- キーワード:マイクロアレイ、データベース、Windows EXCEL
- 担当:東北農研・作物機能開発部・生物工学研究室
- 連絡先:019-643-3514
- 区分:東北農業・生物工学
- 分類:科学・参考
背景・ねらい
マイクロアレイ解析では、多数のcDNAクローンのannotationおよび遺伝子発現データを伴うため、総合的なデータベースが必要である。しかし、総合的データベースのソフトは高価であり、使用方法も煩雑な場合が多い。
そこで、市販のWindows EXCELをプラットホームにし、簡易型データベースを半自動的に作成する方法を考案し、マイクロアレイで得られたデータ解析の補助とする。
成果の内容・特徴
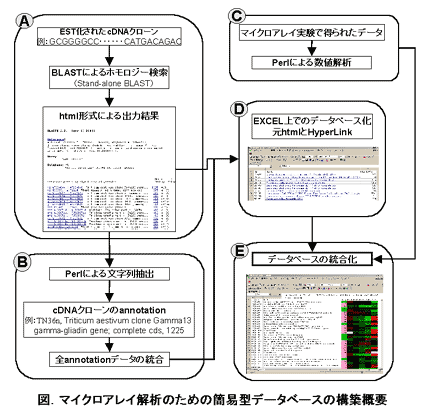
- EST化されたcDNAクローンは、ハードディスクにダウンロードされたデータベースを用いた stand-alone BLASTを用いてホモロジー検索を行い、出力結果はhtml形式で保存する(図-A)。
- html形式の出力結果から最も高い類似度を持つ遺伝子情報をPerlで抽出し、cDNAクローンのannotation とする(図-B)。
- 得られたcDNAクローンのannotationは統合された後、EXCEL上に元のhtmlファイルとHyper-Linkする形でデータベース化する(図-D)。
- マイクロアレイで得られた遺伝子発現データはPerlで処理した後、図-Dで得られたデータベースと統合する。その後、EXCELのマクロを利用しcDNAクローンの類似度及び遺伝子発現データを色分けする(図-C、E)。
- 本データベースは、EXCELを用いた簡易型であるが、汎用性があり自由に仕様を変更できる。また、cDNAクローンの類似度や遺伝子発現データが色分けにより可視化されているため、概観的にデータを捉える事ができる。さらには、cDNAクローンのannotationは、元のhtmlファイルとHyperLinkしているため、クリックするだけで自動的にhtmlファイルを呼び出せ、そのファイルから関連情報をWWWから取得が可能である。
成果の活用面・留意点
- 解析用専用コンピュータを含めた研究資源が不足している比較的小さな研究グループなどでは、解析及び解析方法の一例として有効的に活用できる。
- cDNAクローンの発現分類を行うための統計的手法については、別途検討を要する。
- 半自動化のため、バッチコマンドやPerlに対する知識が必要であり、EXCELが導入されていない場合やOSがMacintoshの場合は別途検討が必要である。
具体的データ
その他
- 研究課題名:コムギ種子形成過程で特異的に発現する遺伝子群の解明
- 予算区分:委託プロ(マイクロアレイ)・パイオニア特別
- 研究期間:1999~2001年度
- 研究担当者:米丸淳一、中村俊樹、齊藤美香、新畑智也((株)日本製粉中央研究所)
